Introduction
Les séquences génomiques complètes de divers organismes, y compris les mammifères, sont récemment devenues disponibles grâce aux progrès rapides de la technologie de séquençage de l’ADN. Cependant, en particulier chez les mammifères, l’analyse des transcrits joue toujours un rôle clé pour combler le fossé entre le génome et le protéome. Ceci est principalement dû au fait qu’à l’heure actuelle, nous ne pouvons pas prédire avec précision les structures des transcrits dérivés d’un gène particulier à partir de la seule information génomique. Ainsi, en tant que méthode d’analyse des transcriptions, la construction d’une bibliothèque d’ADNc est cruciale, même à l’ère du séquençage post-génome. Bien que le clonage d’ADNc de gènes d’intérêt par PCR ait fourni une voie alternative simplifiée pour analyser les structures de transcription sans construction de bibliothèque d’ADNc, la construction d’une bibliothèque d’ADNc est l’approche de choix lorsqu’un grand nombre d’ADNc provenant d’une seule source d’ARNm doivent être analysés. À ce jour, beaucoup d’efforts ont été déployés pour développer une méthode de préparation de bibliothèques d’ADNc de haute qualité (1-4). En revanche, la question de savoir comment préparer une bibliothèque d’ADNc de haute qualité à partir d’un petit bassin d’ARN n’a pas été aussi activement abordée. Cependant, cela est devenu un objectif hautement prioritaire car les chercheurs s’intéressent souvent à des gènes hypothétiques qui ne devraient être exprimés que dans certains types de cellules ou de tissus dans des conditions particulières, telles que celles observées dans des échantillons pathologiques.
Il y a eu un certain nombre de rapports décrivant l’utilisation de petites quantités d’ARN source pour générer des ADNc ou des aRNC amplifiés, à partir desquels des cibles pour l’analyse de microréseaux peuvent être préparées (5-15). Leur objectif final est d’obtenir un ARN de pleine longueur, non biaisé par la population, très représentatif de l’ARNm d’origine. Pour ce faire, ces méthodes adoptent généralement la PCR ou la transcription in vitro par l’ARN polymérase T7 pour amplifier l’ARNm original sous forme d’ADNc ou d’aRNC, respectivement. Bien que la comparaison des profils d’expression soit l’une des approches les plus efficaces pour étudier les différences dans les états physiologiques des cellules ou des tissus, la caractérisation structurelle des transcrits (par exemple, identification de modèles d’épissage alternatifs, site de début de transcription et site de terminaison de transcription) ne peut pas être effectuée par une telle analyse de microréseaux. L’analyse séquentielle de chaque transcription est inévitable dans ces cas.
Nous avons développé une méthode qui permet d’utiliser une petite quantité d’ARN de départ pour construire une banque d’ADNc adaptée au clonage de gènes et à l’analyse complète du séquençage. Le procédé marque les extrémités 3′ et 5′ des ADNC du premier tour avec des séquences promotrices de phages T7 et SP6, respectivement, pour minimiser les effets de biais de taille pendant l’amplification. Après amplification de l’aRNC en deux tours, nous avons converti les ARNC amplifiés en ARNC, cloné ces produits en un plasmide, puis évalué la banque d’ADNc résultante en comparaison avec celle construite par une méthode conventionnelle (4,16,17).
Matériaux et méthodes
Amplification de l’aRNC
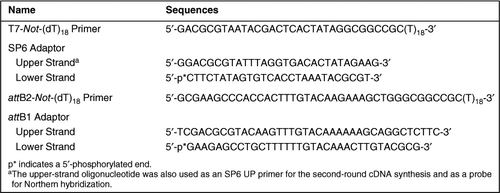
Les séquences d’oligonucléotides utilisées dans cette étude sont présentées dans le tableau 1. Pour établir le protocole, nous avons utilisé 1 µg d’ARN total préparé à partir du cerveau de souris ICR (souris mâles âgées de 8 semaines) comme modèle. Un mélange (10 µL) de 1 µg d’ARN total et de 100 pmol d’amorce T7-Not-(DT)18 a été incubé à 70°C pendant 10 min puis refroidi par encliquetage sur glace. La synthèse d’ADNc double brin et la ligature de l’adaptateur ont été réalisées selon les protocoles du système plasmidique SUPERSCRIPT® (Invitrogen, Carlsbad, CA, USA) (4,16,17), avec des modifications mineures. Brièvement, pour la synthèse de l’ADNc du premier brin, 4 µL de tampon 5× premier brin (Invitrogen), 1 µL de dithiothréitol 0,1 M (DTT), 1 µL de DNTP 10 mM, 1 µL (40 U) RNaseOUT™ (Invitrogen) et 1 µL d’eau ont été ajoutés au mélange d’amorces ARN/T7-Not-(DT)18 dénaturé et incubés à 37 ° C pendant 3 min pour recuire les amorces. Ensuite, 2 µL (400 U) d’EXPOSANT III RNase H-transcriptase inverse (Invitrogène) ont été ajoutés au mélange réactionnel et la température a été ajustée à 50 ° C pour démarrer la synthèse de l’ADNc du premier brin. Après 1 h, la synthèse de l’ADNc du deuxième brin a été réalisée comme décrit précédemment par Gubler et Hoffman (18); au mélange d’ADNc du premier brin, 91 µL d’eau, 30 µL de tampon 5× deuxième brin (Invitrogen), 3 µL de dNTPs 10 mM, 1 µL (10 U) d’ADN ligase d’Escherichia coli, 4 µL (40 U) d’ADN polymérase d’E. coli et 1 µL (2 U) d’E. coli RNase H ont été ajoutés, et le mélange a été incubé à 16°C pendant 2 h. Les terminaisons d’ADNc ont ensuite été polies en bout avec de l’ADN polymérase 10 U T4 (Invitrogen) à 16 °C pendant 5 min, et la réaction a été arrêtée par addition de 10 µL 0,5 M d’acide éthylènediamine tétraacétique (EDTA). Les ADNC obtenus ont été purifiés par extraction avec phénol / chloroforme /alcool isoamylique (25: 24: 1), suivie d’une précipitation à l’éthanol comme décrit précédemment (17), sauf utilisation de 1 µg d’ARNt de levure à la place de l’acide polyadénylique comme support. L’ADNc précipité a été dissous dans 50 µL de TE (Tris-HCl 10 mM, pH 8,0 et EDTA 1 mM), mélangé à 30 µL d’une solution de NaCl à 20% (p/v) de polyéthylène glycol (PEG) 6000/2,5 M, puis incubé sur glace pendant 1 h (17). Après l’incubation, le mélange d’ADNc a été centrifugé à 18 000×g à 4°C pendant 15 min. Le culot d’ADNc obtenu est rincé deux fois avec de l’éthanol à 70%, séché puis remis en suspension dans 30 µL d’eau. Les ADNC purifiés ont été ligaturés avec un adaptateur SP6 de 500 pmol (Tableau 1) en utilisant une ADN ligase 5 U T4 (Invitrogène) dans un volume réactionnel de 50 µL à 16°C pendant une nuit. Le mélange ligaturé par adaptateur a été dilué deux fois avec de l’eau, puis les ADNC ont été purifiés à l’aide d’une colonne DNAclear™ (Ambion, Austin, TX, USA). L’éluat (dans 16 µL d’eau) a été utilisé comme matrice pour la synthèse des aRNC. En utilisant le kit MEGAscript® T7 (Ambion), les ARNC ont été synthétisés à 37 ° C pendant une nuit dans un volume de réaction de 40 µL. Après dégradation des ARNC modèles par 4 U DNase I, les ARNC synthétisés ont été purifiés à l’aide d’un kit RNeasy® Mini (QIAGEN, Valencia, CA, USA) et élués avec 100 µL d’eau. La concentration de l’aRNC a été déterminée par absorption ultraviolette (UV). Pour l’amplification de l’aRNC de deuxième cycle, 2 µg de l’aRNC synthétisé et 100 pmol d’amorce SP6 UP (égale à l’oligonucléotide du brin supérieur de l’adaptateur SP6 indiqué dans le tableau 1) ont été utilisés comme matrice et amorce, respectivement. La synthèse d’ADNc de deuxième tour a été réalisée comme dans la transcription inverse originale à partir de l’ARN source, sauf que le recuit de l’amorce SP6 UP a été effectué à 50 °C pendant 3 min. Après purification de l’ADNc double brin obtenu à travers une colonne DNAclear, 0,5 µg de l’ADNc a été utilisé comme matrice pour la prochaine amplification de l’ADNc assistée par ARN polymérase SP6. En utilisant le kit MEGAscript SP6 (Ambion), la synthèse des aRNC a été réalisée à 37 ° C pendant 6 h. Après dégradation de l’ADNc modèle à l’aide de la DNase I, les ARNC synthétisés ont été purifiés comme décrit ci-dessus.
Construction de la Bibliothèque d’ADNc
Deux microgrammes des ARNC résultants ont été soumis à une synthèse d’ADNc double brin à l’aide d’une amorce attB2-Not-(DT)18 de 100 pmol Tableau 1). Les étapes de synthèse d’ADNc, d’émoussement d’extrémité, de purification et de ligature d’adaptateur ont été effectuées comme dans les synthèses précédentes d’ADNc, sauf que l’adaptateur attB1 (tableau 1 500 pmol) a été ligaturé sur les ADNc double brin. Après purification des ADNC ligaturés par adaptateur attB1 par des traitements successifs d’extraction phénol/chloroforme/alcool isoamylique, de précipitation d’éthanol et de précipitation de PEG/NaCl dans cet ordre, ils ont été dissous dans 50 µL de TE et traités avec de la RNase A (à une concentration finale de 10 µg/mL; Invitrogène) pour dégrader l’ARN contaminé à 37 °C pendant 30 min. Le mélange réactionnel est à nouveau purifié par extraction avec phénol/chloroforme/alcool isoamylique, suivi d’une précipitation éthanol et d’une précipitation PEG/NaCl. Les ADNC résultants ont été dissous dans 15 µL d’TE, et leur quantité a été estimée à partir de l’intensité de coloration fluorescente après électrophorèse sur gel d’agarose (4). Les ADNC ligaturés en attB1 (36 ng) ont été soumis à une réaction de recombinaison in vitro (Système Gateway® ; Invitrogen) avec un vecteur donneur attP-pSP73 de 250 ng comme décrit précédemment (4,16,17). En bref, l’ADNc ligoté attB1 et le vecteur donneur attP-pSP73 ont été mélangés et incubés dans un volume réactionnel de 10 µL contenant 2 µL de tampon réactionnel Clonase™ 5× BP et 2 µL de Clonase BP (tous deux d’Invitrogen) à 25°C pendant une nuit. Le mélange d’ADNc a été traité avec 2 µg de protéinase K (Invitrogène) à 37 ° C pendant 10 min pour éteindre la réaction et extrait avec phénol / chloroforme /alcool isoamylique, suivi d’une précipitation à l’éthanol avec 2 µg d’ARNt de levure comme support. L’ADNc précipité a été dissous dans 10 µL d’eau et 1 µL du mélange a été utilisé pour la transformation des cellules d’E. coli ElectroMAX™ DH10B™ (Invitrogen). Après titrage de la banque d’ADNc résultante, les plasmides d’ADNc ont été récupérés à partir d’environ 1 000 000 colonies par une méthode de dodécylsulfate de sodium alcalin (SDS) telle que décrite précédemment (4,19). Les clones d’ADNc obtenus étaient sous une forme de plasmide portant des sites attL. Les plasmides porteurs de sites attB étant nécessaires dans certaines applications, ces plasmides ont été convertis en ceux porteurs de sites attB comme décrit précédemment (16). Cette bibliothèque d’ADNc a donc été désignée comme MB-AL (bibliothèque dérivée d’aRNC amplifiée par le cerveau de souris).
De la même manière que pour MB-AL, nous avons construit une bibliothèque d’ADNc à partir d’ARN non amplifié (100 µg d’ARN total du cerveau de souris) et l’avons désignée comme MB-CL (bibliothèque de cerveau de souris construite classiquement).
Examen de l’effet de biais de taille sur les ARNC amplifiés
Pour examiner l’effet de biais de taille sur les ARNC, les ARNC du premier tour transcrits par l’ARN polymérase T7 et les ARNC du deuxième tour transcrits par l’ARNC polymérase SP6 ont été exécutés sur gel d’agarose à 1,0% (1 µg chacun) puis épongés sur membrane de nylon (membrane de nylon Biodyne® B; PALL, East Hills, NY, USA). Ils ont été hybridés avec une amorce SP6 UP marquée au 32P (Tableau 1) et un oligonucléotide non-(DT)18 (5′-GCGGCCGCTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTTT-3′), respectivement. Dix picomoles de chaque sonde ont été marqués avec de l’ATP (environ 3000 Ci/mmol; Amersham Biosciences, Piscataway, NJ, États-Unis) en utilisant la polynucléotide kinase T4 (Takara, Kyoto Japon). Après hybridation nocturne dans le tampon GMC (20) à 45°C, ces membranes ont été largement lavées dans une solution de citrate salin standard (SSC) / 1% de SDS trois fois à température ambiante. L’analyse des signaux hybridés a été réalisée sur un Analyseur d’Images BAS 2000 (Fuji Photo Film, Tokyo, Japon)
Évaluation des banques d’ADNc
électrophorèse sur Gel. La distribution des tailles d’insertion d’ADNc a été examinée par électrophorèse des ARNC synthétisés par l’ARN polymérase T7 en utilisant des ARNC plasmidiques MB-AL et MB-CL non digérés comme modèles. Après purification à l’aide d’un Kit RNeasy Mini, les ARNC ont été électrophorés sur un gel d’agarose contenant du formaldéhyde avec des Marqueurs d’ARN Parfaits (EMD Biosciences, Madison, WI, USA). Après coloration du gel avec SYBR® Green II (Invitrogen), l’intensité de coloration fluorescente a été analysée à l’aide du logiciel ImageQuant® (version 5.0) sur un FluorImager 595 (Amersham Biosciences).
Analyse de séquençage aléatoire en un seul passage. Les séquences d’extrémité 3′ des clones d’ADNc ont été examinées par séquençage de l’ADN. L’ADN plasmidique de 768 clones choisis au hasard a été purifié à l’aide d’un Magnia® MFX-9600 (Toyobo, Osaka, Japon) et analysé par un séquenceur capillaire RISA 384 (Shimadzu, Kyoto, Japon) (16). Après avoir rogné la séquence vectorielle à l’aide de la version 4.1 du logiciel Sequencher™ (Logiciel Hitachi, Tokyo, Japon), les séquences d’ADNc obtenues de plus de 200 résidus nucléotidiques ont été regroupées avec des entrées d’ADNc dans la base de données GenBank® et notre base de données de souris interne. L’examen de l’intégrité des extrémités 3′ des clones d’ADNc a été effectué par une analyse de la présence ou non d’un hexamère de signal de polyadénylation canonique (5′-AATAAA-3′) ou de ses variants à base unique (11 hexamères de signal) au sein des 50 résidus nucléotidiques en amont de la queue poly(A) des ADNc (21).
Microarray d’ADNc. Des analyses de microréseaux ont été effectuées pour comparer les populations d’ADNc dans les bibliothèques MB-AL et MB-CL. Des microréseaux de nylon ADNc faits maison avec 3534 sondes ont été préparés et une détection radio-isotopique a été effectuée (22). Brièvement, des plasmides d’ADNc, qui ont été isolés dans notre institut et pour lesquels des séquences complètes ou terminales étaient déjà connues, ont été repérés à l’aide de GeneTAC™ RA1 (Genomic Solutions, Ann Arbor, MI, USA) sur membrane de nylon Biodyne B, puis fixés en suivant les instructions du fabricant. La liste des ADNC immobilisés sur cette puce est disponible sur demande auprès des auteurs. Les ADNc cibles MB-AL et MB-CL ont été préparés en utilisant la transcription inverse de chacun des échantillons d’aRNC de 1 µg utilisés pour l’analyse de la taille de l’insert comme décrit ci-dessus. Ces cibles ont été synthétisées et marquées dans le mélange réactionnel contenant 1 µg de chaque aRNC, 100 ng d’hexamère aléatoire, 1 × tampon de premier brin, 10 mm DTT, chaque 800 µM dATP, dTTP et dGTP, 800 nM dCTP, 5 µL dCTP (> 2500 Ci/mmol; Amersham Biosciences), et 100 U exposant II RNase H-inverse transcriptase à température ambiante pendant 10 min puis à 42°C pendant 1 h. Après lyse alcaline et neutralisation, les ADNC marqués ont été purifiés avec le kit de purification QIAquick® PCR (QIAGEN) et comptés. L’ADNc cible a été soumis à une hybridation avec la puce nylon ADNc en présence de 1 µg d’acide polyadénylique et de 2,5 µg d’ADN Cot-1 de souris® (Invitrogen) dans du tampon PerfectHyb™ (Toyobo) de 250 µL à 68°C pendant une nuit. Après un lavage rigoureux à 68°C avec 2× SSC/1% SDS (deux lavages de 15 min chacun) puis avec 0,1× SSC/1% SDS (deux lavages de 30 min chacun), les signaux d’hybridation ont été détectés et analysés sur un FLA-8000 (film photo Fuji). Les taches d’ADNc présentant des signaux plus forts que ceux du témoin de fond ont été sélectionnées et soumises à une analyse plus poussée. Après avoir effectué une normalisation globale, des diagrammes de dispersion des intensités de signal de taches d’ADNc provenant de cibles MB-AL et MB-CL ont été obtenus.
Hybridation par transfert d’ARN. L’hybridation par transfert d’ARN a été réalisée pour examiner les effets du biais de taille. Les ARNC MB-AL et MB-CL (1,5 µg chacun, synthétisés comme décrit ci-dessus) ont été électrophorés sur gel d’agarose contenant du formaldéhyde et transférés sur membrane de nylon Biodyne B. Les ADNC de la sonde ont été préparés par amplification PCR. Les amorces ont été conçues à partir des séquences enregistrées dans la base de données GenBank ou dans notre base de données interne; les modèles étaient les plasmides d’ADNc que nous avions isolés. La sonde de la protéine S6 ribosomique avait une longueur de 724 pb, amplifiée avec des amorces de 5′-CGCTCGGCTGTGTCAAGATG-3′ et 5′-GAGGACAGCCTACGTCTCTTGG-3′, et la sonde de la protéine de choc thermique (hsp) de 70, 940 pb de longueur, a été amplifiée avec 5′-GATGGACAAGGCGCAGATCC-3′ et 5′-CTCGATGGTGGGTCCTGAGC – amorces de 3′. Ces sondes ont été marquées avec du dCTP (environ 3000 Ci / mmol; Amersham Biosciences) à l’aide du système de marquage de l’ADN RadPrime (Invitrogen). Après hybridation nocturne en tampon PerfectHyb à 65°C, les membranes ont été lavées successivement avec 0,1× SSC/1% de SDS à température ambiante pendant 5 min et pendant 15 min, puis à 65°C pendant 30 min. Les signaux d’hybridation ont été détectés sur un analyseur d’images BAS 2000.
Résultats et discussion
La méthode de construction de la bibliothèque d’ADNc que nous avons développée dans cette étude est illustrée à la figure 1 (les étapes nouvellement introduites sont mises en évidence par une étoile). Cette méthode consiste en deux cycles d’étape d’amplification des aRNC, de conversion des ARNC en ARNC et de clonage recombinationnel en un plasmide. L’étape clé de cette méthode est la ligature de l’adaptateur pour marquer les extrémités de l’ADNc avec la séquence promotrice SP6. Une fois que nous pouvons synthétiser avec succès les ADNC dans ce format, il devient possible de transcrire spécifiquement les ARNC du premier tour qui contiennent la séquence promotrice SP6 à leur extrémité 3 ‘. Les ARNC de deuxième cycle sont synthétisés avec l’ARN polymérase SP6 en utilisant les ARNC double brin résultants comme modèles. Après les deux cycles d’amplification assistée par ARN polymérase, les ARNC résultants sont ensuite convertis en ARNC double brin par transcription inverse à l’aide de l’amorce attB2-Not-(DT)18. L’utilisation de l’amorce attB2-Not-(DT)18 dans la transcription inverse nous permet de convertir uniquement les ARNC portant la séquence 5′-(A)18GCGGCCGC-3′ à leurs extrémités 3’. Étant donné que ces modifications ne devraient permettre que l’amplification et le clonage d’ADNC ayant des extrémités correctement marquées, nous nous attendons à ce que cette méthode minimise le biais de taille lors de l’amplification assistée par ARN polymérase.

Les étapes de l’amplification de l’aRNC assistée par ligature par adaptateur et la construction de la bibliothèque d’ADNc suivante sont illustrées. Pour maintenir les tailles d’ADNc amplifiées avec celles des ADNc du premier tour, nous avons introduit quelques modifications dans les méthodes conventionnelles assistées par l’ARN polymérase (mises en évidence par des étoiles). Les lignes bleues et rouges indiquent respectivement l’ADNc et l’ARN. E. coli, Escherichia coli.
Pour tester l’efficacité de la méthode, nous avons construit des banques d’ADNc par une méthode conventionnelle (16) en utilisant de l’ARN total non amplifié provenant du cerveau de souris (100 µg) et par la méthode décrite ci-dessus en utilisant 1 µg d’ARN total provenant du cerveau de souris. Le tableau 2 résume les quantités d’ADNc et d’aRNC obtenues à chaque étape, calculées comme une moyenne de trois essais expérimentaux indépendants de construction MB-AL. Nous avons obtenu en moyenne 6,4×107 clones d’ADNc indépendants comme sortie finale. Étant donné que ce nombre de clones d’ADNc provient d’un total de 36 ng d’ADNc générés à l’aide du protocole d’amplification, nous calculons que cette méthode donne environ 1,2 ×1011 clones d’ADNc à partir de 1 µg d’ARN total.

Pour examiner expérimentalement l’effet de biais de taille lors des étapes d’amplification, nous avons ensuite comparé les distributions de taille de l’aRNC du premier et du deuxième tour par analyse d’hybridation par transfert d’ARN. Dans ces expériences, les ARNC du premier tour et du deuxième tour ont été hybridés avec l’amorce SP6 UP et avec l’oligonucléotide Non-(dT) 18, respectivement, car ces sondes oligonucléotidiques ne détectent que les ARNC correctement transcrits jusqu’à la fin. Comme le montre la figure 2, la taille de l’ARN au pic du signal d’hybridation dans l’aRNC du deuxième tour s’est avérée presque identique à celle de l’aRNC du premier tour, mais avec un léger décalage vers une taille plus petite. Nous soupçonnons que la troncature des ARNC a probablement eu lieu lors de la synthèse de l’ADNc de deuxième cycle avec l’amorce SP6 UP.

Une hybridation par transfert d’ARN a été réalisée pour examiner l’effet de biais de taille sur les ARNC. Les signaux d’hybridation suivant la direction de l’électrophorèse de l’aRNC ont été tracés. La taille de l’ARN a été déterminée en fonction de la mobilité du marqueur d’échelle d’ARN. Ligne bleue, l’aRNC du premier tour hybridé avec la sonde SP6 UP; ligne rouge, l’aRNC du deuxième tour hybridé avec la sonde Not-(dT) 18; Valeur PSL, intensité du signal indiquée arbitrairement par le Logiciel de mesure d’image (Film photo Fuji).
Pour évaluer davantage la qualité de la bibliothèque d’ADNc amplifiée résultante, nous avons également comparé diverses caractéristiques des bibliothèques d’ADNc générées avec et sans l’amplification (MB-AL et MB-CL, respectivement). Nous avons d’abord examiné la distribution des tailles des inserts d’ADNc, comme le montre la figure 3A. Les résultats des signaux d’intensité de fluorescence obtenus à partir de l’image de coloration sur gel indiquaient que les tailles des inserts d’ADNc de MB-AL et de MB-CL étaient similaires, mais le point de crête était légèrement plus petit dans MB-AL (0,65 kb) par rapport à celui dans MB-CL (0,8 kb). Ensuite, nous avons analysé des séquences en 3′ de clones d’ADNc échantillonnés aléatoirement de chaque bibliothèque. Les résultats de regroupement pour ces étiquettes de séquence exprimées (EST) sont présentés à la figure 3B et suggèrent que la complexité de MB-AL était aussi élevée que celle de MB-CL. Cependant, il convient de noter que les clones d’ADNc hautement redondants ont disparu dans MB-AL. Par une analyse statistique (test Chi), il a été démontré que la différence de redondances entre MB-AL et MB-CL était significative (la probabilité que les redondances dans MB-AL et MB-CL soient similaires est < 0,001). De plus, nous avons examiné l’intégrité des extrémités 3′ des ADNC en analysant si chaque EST contenait un hexamère de signal de polyadénylation ou non. Les résultats ont indiqué que 80,1 % et 87,3 % des clones d’ADNc dans MB-AL et MB-CL, respectivement, contenaient des séquences de signaux de polyadénylation plausibles (21). La diminution du taux d’occurrence des hexamères de signal de polyadénylation dans le MB-AL indique probablement une augmentation du nombre de clones d’ADNc où la synthèse d’ADNc a été amorcée en interne en raison de deux cycles d’amorçage dT.

MB-AL (bibliothèque dérivée de cRNA amplifiée par le cerveau de souris) a été évaluée en comparaison avec une bibliothèque de construction conventionnelle, MB-CL. A) Comparaison des longueurs d’ADNc de l’insert. Les ARNC transcrits in vitro ont été fractionnés en taille sur gel d’agarose et colorés. Leurs intensités fluorescentes le long de la direction de l’électrophorèse (indiquées par la taille de l’ARN) sont représentées en unités fluorescentes relatives (UFR). (B) Résultats de regroupement des séquences d’extrémité MB-AL et MB-CL 3′ (EST). C) Comparaison de la population d’ADNc dans MB-AL et MB-CL. Les signaux obtenus par hybridation de microréseaux ont été tracés. (D) Résultats d’hybridation par transfert d’ARN sur la protéine ribosomique S6 et la protéine hsp 70. Les ARNM MB-AL étaient dans la voie de gauche et ceux de MB-CL étaient dans la voie de droite (indiqués par AL et CL, respectivement). Les pointes de flèches indiquent les positions de la taille d’ARN prévue de chaque gène. hsp, protéine de choc thermique.
Par hybridation de microréseaux, nous avons examiné la population d’ADNc de MB-AL en comparaison avec celle de MB-CL. La figure 3C montre un diagramme de dispersion des signaux d’hybridation des cibles MB-AL et MB-CL. Les résultats ont indiqué une corrélation considérablement élevée des signaux d’hybridation entre les cibles MB-AL et MB-CL, suggérant qu’il n’y avait pas d’effet néfaste de l’amplification dans la population d’ADNc.
Enfin, nous avons effectué une analyse d’hybridation par transfert d’ARN pour examiner les effets de biais de taille en portant une attention particulière aux gènes de ménage (Figure 3D). Pour la protéine ribosomique S6 et la protéine hsp 70, les tailles d’aRNC dans MB-AL et MB-CL étaient similaires et comparables à celles attendues de leurs séquences nucléotidiques rapportées. Cependant, le modèle d’hybridation des ARNC hsp 70 chez MB-AL était légèrement différent de celui chez MB-CL, ce qui indique que l’effet de biais de taille n’a pas été complètement éliminé.
La méthode de construction de la bibliothèque d’ADNc décrite dans ce rapport a été conçue pour minimiser les biais de taille lors des étapes d’amplification assistées par l’ARN polymérase. Une telle méthode de construction de bibliothèque assistée par ARN polymérase à partir d’un petit pool d’ARN pour une analyse de séquençage complète n’a pas été bien étudiée, tandis que des méthodes de construction de bibliothèque ADNc/aRNC pour l’hybridation de microréseaux ont été activement poursuivies. Bien que Lukyanov et al. (23) et Piao et coll. (24) méthodes rapportées utilisant l’amplification par PCR pour construire une banque d’ADNc à partir d’une quantité submicrogramme d’ARN total, l’amplification par PCR présente intrinsèquement des inconvénients tels qu’un biais sévère de taille et de population et une faible fidélité de l’amplification par ADNc. C’est pourquoi nous avons essayé de développer une méthode de construction de bibliothèque d’ADNc basée sur l’amplification assistée par ARN polymérase dans cette étude.
Dans ce but, nous avons conçu une nouvelle stratégie d’amplification assistée par ARN polymérase comme le montre la figure 1 car les méthodes d’Eberwine et al. (5), Huang et coll. (10), et Lin et coll. (11) présentent encore des inconvénients pour la préparation d’un ADNc contenant un plasmide adapté à une analyse complète de la structure des gènes. Dans le premier cas, la synthèse d’ADNc après amplification de l’aRNC se fait avec des hexamères aléatoires, ce qui rend les ADNc amplifiés inévitablement plus courts que ceux d’origine (5,7). Dans ce dernier cas, l’introduction d’une queue homopolymérique à l’extrémité de l’ADNc du premier brin par une activité de transcriptase inverse (10,11) de la désoxynucléotidyl transférase terminale (TdT) ou du virus de la leucémie murine Moloney (MMLV) est logique pour minimiser l’effet de biais de taille lors de l’amplification assistée par l’ARN polymérase comme dans notre méthode. Cependant, la méthode de résidus homopolymériques est connue pour provoquer une troncature des ADNc en raison d’un amorçage inattendu de la synthèse d’ADNc à partir d’un étirement homopolymérique de séquences d’ARN internes. Étant donné que la méthode de ligature de l’adaptateur décrite dans cette étude pourrait fournir une séquence spécifique pour l’amorçage de l’ADNc, nous pourrions nous attendre à réduire l’étendue de la troncature de l’ADNc en utilisant notre méthode.
En principe, notre méthode ne devrait amplifier que les ADNc flanqués d’une queue poly(A) intrinsèque et de la séquence adaptatrice SP6, qui ont été générés dans la synthèse d’ADNc du premier tour. Les résultats présentés à la figure 2 et à la figure 3A étaient essentiellement cohérents avec cela, bien que la population d’ADNc dans la plage de taille plus petite ait légèrement augmenté. Les résultats de l’analyse par transfert d’ARN (Figure 3D) ont également montré que l’effet de biais de taille pouvait être réduit mais pas complètement supprimé, même dans notre méthode. Ceci est probablement dû au fait que des ADNc tronqués ajoutés par une queue poly(A) et la séquence promotrice SP6 ont été générés lors de la conversion de l’aRNC en ADNc. Ce point de vue était étayé par le fait que le taux d’occurrence des séquences de signaux de polyadénylation dans MB-AL était significativement plus faible que celui observé dans la banque d’ADNc classique (21). La troncature des ADNC pourrait se produire pendant la synthèse des ADNc en raison de l’amorçage interne de l’ARN avec l’amorce à queue dT et l’amorce SP6. Étant donné que les étirements homopolymériques se produisent fréquemment dans les ARNM eucaryotes, la méthode de tailing homopolymérique pourrait imposer un biais de taille plus sévère sur l’amplification des aRNC à plusieurs tours que notre méthode de ligature adaptative. Bien que l’augmentation de la température de réaction lors de la transcription inverse et la diminution de la concentration de l’amorce puissent supprimer dans une certaine mesure l’amorçage interne aberrant dans la synthèse de l’ADNc, il est connu qu’il est difficile de l’éliminer complètement à l’heure actuelle. Ainsi, bien que nous ayons effectué une amplification cRNA à deux tours pour la démonstration du protocole global, en pratique, nous recommandons de sauter l’amplification cRNA de deuxième tour lorsque suffisamment de cRNA peuvent être obtenus dans la réaction de premier tour.
Compte tenu de ces résultats, nous concluons que notre nouvelle méthode est utile pour la construction d’une bibliothèque d’ADNc à partir d’une petite quantité d’ARN de départ. En fait, nous avons utilisé avec succès et de manière routinière cette méthode pour la construction de banques d’ADNc lorsque la quantité d’ARN total est inférieure à 1 µg (données non représentées). Étant donné que 1 µg d’ARN total pourrait être récupéré à partir de 105-106 cellules de mammifères ou de 1 mg de tissus de mammifères, des banques d’ADNc peuvent être facilement construites à partir de cellules fractionnées par des trieurs de cellules ou une microdissection en utilisant notre méthode. De plus, comme nous calculons que cette méthode nous permet d’obtenir plus de 105 clones d’ADNc à partir d’ARN total de 1 pg, ce qui est proche de la quantité d’ARN total dans une seule cellule, une banque d’ADNc dérivée d’une seule cellule pourrait être construite sur la base de cette amplification d’aRNC assistée par ligature adaptatrice.
Remerciements
Les auteurs sont reconnaissants pour les instructions d’analyse statistique du Dr Kazuharu Misawa, pour la division des ADN plasmidiques du Dr Hisashi Koga et pour l’aide technique de Mme Akiko Ukigai-Ando et de M. Takashi Watanabe. L’étude a été soutenue par une subvention de l’Institut de recherche sur l’ADN de Kazusa à R.O., R.F.K. et O.O.
Déclaration d’intérêts concurrents
Les auteurs ne déclarent aucun intérêt concurrent.
- 1. Il s’agit d’une espèce de plantes herbacées de la famille des » Cyprinaceae » et de la sous-famille des » Cyprinaceae « .. 1996. Clonage d’ADNc pleine longueur à haute efficacité par trappeur à CAPUCHON biotinylé. Génomique 37:327-336.Crossref, Medline, CAS, Google Scholar
- 2. Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « . 1997. Construction et caractérisation de banques d’ADNc du cerveau humain adaptées à l’analyse de clones d’ADNc codant pour des protéines relativement grandes. ADN Rés. 4:53-59.Crossref, Medline, CAS, Google Scholar
- 3. Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » pooideae « , sous-famille des » Pooideae « , originaire d’Asie du Sud-Est. 1997. Construction et caractérisation d’une bibliothèque d’ADNc enrichie sur toute la longueur et d’une bibliothèque d’ADNc enrichie en extrémité 5′. Gène 200: 1149-1156.Crossref, Google Scholar
- 4. Ohara, O. et G. Temple. 2001. Construction d’une banque d’ADNc directionnelle assistée par la réaction de recombinaison in vitro. Acides nucléiques Res. 29:e22.Crossref, Medline, CAS, Google Scholar
- 5. Eberwine, J., H. Yeh, K. Miyashiro, Y. Cao, S. Nair, R. Finnel, M. Zettel et P. Coleman. 1992. Analyse de l’expression des gènes dans des neurones vivants uniques. Proc. Natl. Acad. Sci. États-Unis 89:3010-3014.Crossref, Medline, CAS, Google Scholar
- 6. Il s’agit de l’un des plus importants ouvrages de l’histoire de la marine. 2000. Amplification d’ARNm haute fidélité pour le profilage des gènes. NAT. Biotechnol. 18:457–459.Crossref, Medline, CAS, Google Scholar
- 7. Baugh, L.R., A.A. Hill, E.L. Brown et C.P. Hunter. 2001. Analyse quantitative de l’amplification de l’ARNm par transcription in vitro. Acides nucléiques Res. 29:e29.Crossref, Medline, CAS, Google Scholar
- 8. Zhumabayeva, B., L. Diatchenko, A. Chenchik et P.D. Siebert. 2001. Utilisation de l’ADNc généré par SMART™ pour des études d’expression génique dans plusieurs tumeurs humaines. BioTechniques 30:158-163.Lien, CAS, Google Scholar
- 9. Gomes, L., R. Silva, B.S. Stolf, E.B. Cristo, R. Hirata, Jr., F.A. Soares, L. Reis, E.J. Neves, et al.. 2003. Analyse comparative de l’ARN amplifié et non amplifié pour l’hybridation dans un microarray d’ADNc. Anal. Biochem. 321:244–251.Les résultats de cette étude sont les suivants : Il y a une grande variété de plantes et de plantes. 2003. Bcl-2 tronquée, un marqueur pré-métastique potentiel dans le cancer de la prostate. Biochem. Biophys. Rés. Commun. 306:912–917.Les résultats de cette étude sont les suivants : Lin, S.L. et S.Y. Ying. 2003. Construction d’une bibliothèque d’ARNm/ADNc à l’aide d’une réaction de cycle ARN-polymérase. Méthodes Mol. Biol. 221:129–143.Medline, CAS, Google Scholar
- 12. Matz, M.V. 2003. Amplification of representative cDNA pools from microscopic amounts of animal tissue. Méthodes Mol. Biol. 221:103–116.Medline, CAS, Google Scholar
- 13. Il s’agit de l’une des principales sources d’approvisionnement en eau potable de la région. 2003. Amplification globale de l’ARNm par PCR à commutation de modèles: linéarité et application à l’analyse de microréseaux. Acides nucléiques Res. 22: e142.Crossref, Google Scholar
- 14. Polacek, D.C., A.G. Passerini, C. Shi, N.M. Francesco, E. Manduchi, G.R. Grant, S. Powell, H. Bischof, et al.. 2003. Fidelity and enhanced sensitivity of differential transcription profiles following linear amplification of nanogram amounts of endothelial mRNA. Physiol. Génomique 13:147-156.Les résultats de cette étude sont les suivants : Wilson, C., S.D. Pepper, Y. Hey et C.J. Miller. 2004. Les protocoles d’amplification introduisent des erreurs systématiques mais reproductibles dans les études d’expression génique. BioTechniques 36:498-506.Lien, CAS, Google Scholar
- 16. Ohara, O., T. Nagase, G. Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « .. 2002. Caractérisation de la banque d’ADNc fractionnés en taille générée par la méthode assistée par recombinaison in vitro. ADN Rés. 9:47-57.Les résultats de cette étude sont les suivants : Ohara, O. 2003. Construction d’une banque d’ADNc fractionnée en taille assistée par une réaction de recombinaison in vitro. Méthodes Mol. Biol. 221:59–71.Medline, CAS, Google Scholar
- 18. Gubler, U. et B.J. Hoffman. 1983. Une méthode simple et très efficace pour générer des bibliothèques d’ADNc. Gène 25:263-269.Les résultats de cette étude sont les suivants : Sambrook, J. et D.W. Russell. 2001. Préparation de l’ADN plasmidique par lyse alcaline avec SDS, p. 1.31-1.42. Clonage moléculaire, (3e édition). Presse de laboratoire CSH, Cold Spring Harbor, NY.Google Scholar
- 20. Église, G.M. et W. Gilbert. 1984. Séquençage génomique. Proc. Natl. Acad. Sci. États-Unis 81: 1991-1995.Il s’agit d’un programme de recherche et de recherche sur les technologies de l’information et de la communication. Beaudoing, E., S. Freier, J.R. Wyatt, J.M. Claverie et D. Gautheret. 2000. Patterns of variant polyadenylation signal utilisation in human genes. Genome Res. 10:1001-1010.Les résultats de cette étude sont les suivants : Tanaka, T.S., S.a. Jaradat, M.K. Lim, G.J. Kargul, X. Wang, M.J. Grahovac, S. Pantano, Y. Sano, et al.. 2000. Profilage d’expression à l’échelle du génome du placenta et de l’embryon à mi-gestation à l’aide d’une puce de développement d’ADNc de 15 000 souris. Proc. Natl. Acad. Sci. États-Unis 97:9127-9132.Les résultats de cette étude sont les suivants : Lukyanov, K., L. Diatchenko, A. Chenchik, A. Nanisetti, P. Siebert, N. Usman, M. Matz et S. Lukyanov. 1997. Construction de banques d’ADNc à partir de petites quantités d’ARN total en utilisant l’effet de PCR par supression. Biochem. Biophys. Rés. Commun. 230:285–288.Les résultats de cette étude sont les suivants : Piao, Y., N.T. Ko, M.K. Lim et M. Ko. 2001. Construction de bibliothèques d’ADNc enrichies de transcriptions longues à partir de quantités submicrogrammes d’ARN totaux par une méthode d’amplification par PCR universelle. Genome Res. 11:1553-1558.Crossref, Medline, CAS, Google Scholar
