
Automatisierte DNA-Sequenzierung durch kapillare elektrophorese
Die Trennung der Produkte einer radioaktiv markierten oder fluoreszierenden Didesoxysequenzierungsreaktion wurde in der Vergangenheit in einem Polyacrylamid-Slab-Gel durchgeführt, wie es beispielsweise in einem 96-Lane ABI 377 verwendet wird. Zu den Nachteilen dieses Verfahrens gehören die Notwendigkeit, für jeden Sequenzierungslauf ein frisches Gel herzustellen, das manuelle Laden von Proben in das Gel, relativ lange Laufzeiten (8 bis 10 Stunden für die Auflösung eines 1-KB-DNA-Fragments) und Schwierigkeiten bei der automatisierten und / oder manuellen Verfolgung von Spuren.
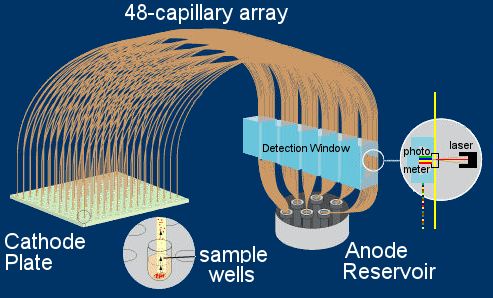
Eine alternative Trennmethode ist die Kapillarelektrophorese. Eine Reihe von Standard-Dideoxy-DNA-Sequenzierungsreaktionen werden jeweils in einem Well einer 8-Zeilen- x 12-Spalten-96-Well-Platte hergestellt. Eine Reihe von ultradünnen Kapillarröhrchen (0.1 mm Innenbohrung, 50 ~ 80 cm Länge) werden mit einer Harzperlenmischung gefüllt und in ein Multi-Kapillararray gegeben. Ein Ende des Arrays ist in einer negativ geladenen Kathodenplatte montiert, so dass ihre Enden mit jeder Vertiefung der Probenplatte fluchten. Das Anlegen von Hochspannung bewirkt, dass die markierte DNA in jedem Probenbrunnen in die entsprechende Kapillare eintritt und durch sie in Richtung des positiv geladenen Anodenreservoirs wandert. Wegen der Hochspannungen verwendet, elektrophoretische Trennung erfordert nur 1 ~ 3 Stunden. Wie bei der Slab-Gel-Elektrophorese bewegen sich die kleineren Fragmente schneller als die größeren . Die Endmarkierungsfarbstoffe werden durch einen Laser im Detektorfenster aktiviert , und die Fluoreszenzwellenlänge jedes Fragments wird von einem Photometer abgelesen, wenn es einen festen Punkt passiert. Im Gegensatz zum Slab-Gel-System sind Laser und Photometer fest und unbeweglich und können mehrere nebeneinander liegende Kapillaren gleichzeitig als separate Datenströme aktivieren und lesen. Mobilitätsdaten werden an einen Computer gesendet, und für jede Probe wird ein Chromatogramm erzeugt, das im Wesentlichen mit denen des Plattengelsystems identisch ist.
Kapillarsequenzierer verwenden typischerweise einen Roboter, um einen Stapel von Probenplatten automatisch zu laden. Die Kapillaranordnung wird mit Puffer zwischen den Platten gespült, so dass die Maschine für viele Läufe unbeaufsichtigt laufen gelassen werden kann. In den größten Einrichtungen können PCR-Reaktionen auch direkt mit Sequenzierplatten in Roboterarbeitsplätzen verknüpft werden, die Dutzende oder Hunderte von Kapillarsequenzierern bedienen. Die Chromatogrammdaten werden auf einem Server abgelegt und können von Benutzern heruntergeladen werden, die Hunderte oder Tausende von Kilometern entfernt sind. Skaleneffekte können es billiger machen, DNA-Sequenzierung auszulagern, als eine interne Maschine zu kaufen, zu warten und zu besetzen.
