de ce dezvolta analiza capac de exprimare a genelor?
cercetările efectuate De Piero Carninci și Yoshihide Hayashizaki la sfârșitul anilor 1990, care au început cu metoda cap trapper, utilizarea trehalozei, metoda de normalizare/scădere și un nou vector de clonare, au stabilit scena pentru dezvoltarea analizei Cap a expresiei genelor. Cu cap trapper, hibrizii ADNc/ARNm de lungime întreagă sunt izolați, iar ARNm este biotinilat chimic pe structura capacului, iar margelele magnetice acoperite cu streptavidină captează hibrizii. Acest avans în seria de tehnologii ADN majore este descris de natură ca Milestone 5 (www.nature.com/milestones/miledna/full/miledna05.html). scopul lor în dezvoltarea CAGE a fost de a crea o tehnologie pentru cartografierea cuprinzătoare a marea majoritate a site-urilor de pornire a transcrierii umane și a promotorilor acestora. De fapt, tehnologia de profilare a activității transcrierii genei la fiecare site promotor nu a existat până când CAGE a venit pe scenă. ARN-ul mesager (ARNm) reprezintă o legătură critică între informațiile codificate în genele individuale ale unui genom și structura proteică care determină soarta unui organism. Ne-am întrebat: care sunt regiunile genomice sau promotorii care determină expresia specifică a genelor și ARN-urile lor unice diferite? Într-adevăr, colecțiile ADNc de lungă durată au arătat că majoritatea genelor au mai mult de un loc de pornire a transcrierii și, prin urmare, este destul de dificil să se identifice regiunile de control, adică promotorii, responsabili de exprimarea diferitelor forme de transcrieri. analiza complexității acestui proces de transfer de informații, numit ‘transcriere’, necesită dezvoltarea unor instrumente moleculare sofisticate care pot capta atât aspectele calitative, cât și cantitative ale expresiei genelor. Deci, cu CAGE, tehnologia noastră originală de detectare a transcrierii la nivel de genom, putem face profilarea expresiei genelor cu identificarea simultană a siturilor de pornire transcripționale specifice țesutului/celulei/stării (TSS), inclusiv analiza utilizării promotorului. CAGE se bazează pe prepararea și secvențierea concatamerilor de etichete ADN derivate din nucleotidele inițiale 20 de la ARNm de capăt 5′, care reflectă concentrația inițială a ARNm în proba analizată (frecvența ARN).
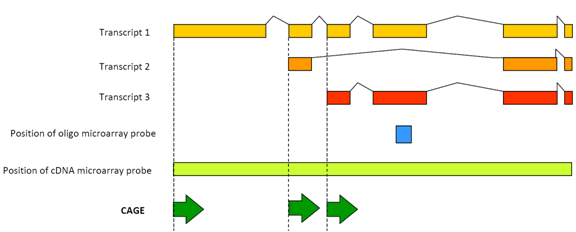
Figura 1: CAGE detectează activitatea transcripțională a fiecărui promotor transcriere.
etichetele de secvențiere exprimate (Est) au fost utilizate în stadiile incipiente ale dezvoltării tehnologiei pentru a identifica elementele promotorului prin alinierea lor la genomul uman. Cu toate acestea, acest proces este foarte scump din cauza costurilor de manipulare a CDN-urilor fizice și a secvențierii Sanger. O modalitate de a depăși aceste probleme este utilizarea tehnologiilor de etichetare, care au fost dezvoltate pentru a detecta transcrierile cu o sensibilitate care este cu cel puțin un ordin de mărime mai mare decât secvențierea EST, apoi identificarea exhaustivă a transcrierilor, identificarea promotorilor lor și corelarea acestora cu profilurile de Expresie prin numărarea etichetelor ca măsură digitală a expresiei genelor.
aceste secvențe (numite și „etichete”) sunt apoi aliniate la secvențele genomului prin proceduri computaționale simple (numite explozie) și numărate, ceea ce oferă o măsurare a frecvenței expresiei ARN. Pe măsură ce aceste etichete de secvență identifică locurile de pornire ale transcripției ARN, ele identifică și secvențele genomului apropiate de acele locuri de pornire. Regiunile învecinate sunt regiunile promotor de bază, care sunt secvențe genomice care determină transcrierea genelor la numeroasele condiții diferite care se întâlnesc în multe organisme complexe, de la șoareci la oameni.
atributele CAGE
CAGE are avantaje mari în comparație cu tehnicile clasice de detectare a expresiei bazate pe microarray. De fapt, prin identificarea promotorului care provoacă transcrierea ARN în fiecare fenomen biologic, țesut, celulă etc. putem identifica elementele de reglare a ADN-ului specifice fiecărui fenomen biologic, analizând secvențele care se află în promotorii izoformelor ARN exprimate în probele analizate. Promotorii conțin secvențe specifice sau site-uri de legare a factorului de transcripție (TFBS), care sunt recunoscute de proteinele lor de legare numite factori de transcripție (TF) și promovează sau, alternativ, reprimă transcripția. Folosind Metode computaționale, cercetătorii noștri analizează promotorii care au profiluri de Expresie similare pentru TFB-urile lor și apoi identifică TFs responsabil pentru ieșirea transcripțională a genomului. Prin numărarea numărului de etichete de cușcă pentru fiecare promotor din cadrul unei gene, putem determina acum nu numai nivelul de Expresie ARN (aceasta este o detectare digitală a frecvenței), ci, important, și din care dintre diferiții promotori alternativi ARN este transcris.
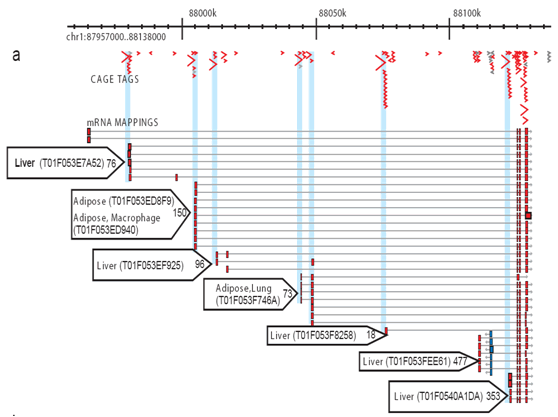
Figura 2: CAGE permite profilarea completă a activărilor la fiecare site promotor. Pentru fiecare bibliotecă, un număr de etichete de cușcă sunt secvențiate și aliniate la genom, astfel încât activitatea transcripțională specifică la fiecare promotor să poată fi măsurată și contribuția fiecărui promotor să se distingă. Acest exemplu simplificat arată promotorii de bază adipos și ficat. Săgeți albastre mici: etichete individuale cuști; săgeți roșii: preferința de utilizare a promotorului pentru țesuturi; cutii roșii: regiuni promotor de bază.
după cum sa menționat, CAGE folosește captarea capacului ca prim pas pentru a captura capetele 5′ ale ADNc, care sunt apoi transformate în secvență scurtă (etichete) de 20 până la 27 nt corespunzătoare ARNm TSSs, . Am produs milioane de etichete de cușcă pentru șoareci și oameni folosind etichete de cușcă concatenate cu secvențiere Sanger, până când ne-am mutat recent în deepCAGE, pentru care folosim secvențierea de a doua generație.
până în 2006 am fost difuzate biblioteci cușcă pe conducta noastră de secvențiere RISA originală, care a fost construit la sfârșitul anilor ‘ 90 și a inclus singurul sequencer capilar cu o serie de 384 capilare pe care le-am dezvoltat în colaborare cu Shimadzu Corporation.
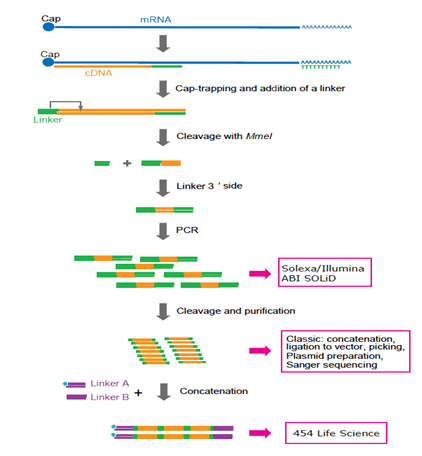
Figura 3: reprezentarea Protocolului de pregătire a cuștilor adaptat diferitelor platforme. Acum sunt preferate Solexa și Illumina. 454 Life Sciences (sistemul FLX) nu mai este folosit deoarece concatenarea necesită cicluri PCR suplimentare și manipulare complicată. În viitor, tehnologia de secvențiere cu o singură moleculă va fi preferată, deoarece este posibil ca PCR să nu fie necesară.
deși tehnologia de etichetare și secvențiere a fost dezvoltată folosind metoda Sage (serial analysis of gene analysis), CAGE este unică deoarece se bazează pe principiul profilării capătului 5′ al ARN-urilor care poartă un ARNm – sit-toate ARN-urile și o mare parte din ARN-urile necodificate. Am dezvoltat etichete cu cușcă lungă de 27 nt pentru a crește eficiența cartografierii.
