Introduction
de volledige genoomsequenties van verschillende organismen, waaronder zoogdieren, zijn onlangs beschikbaar gekomen als gevolg van snelle vooruitgang in DNA sequencing technologie. Nochtans, in het bijzonder in zoogdieren, speelt de analyse van afschriften nog een belangrijke rol in het overbruggen van het hiaat tussen het genoom en proteome. Dit is vooral omdat, op dit moment, kunnen we niet nauwkeurig voorspellen de structuren van transcripten afgeleid van een bepaald gen uit de genomische informatie alleen. Aldus, als methode voor de analyse van afschriften, is de bibliotheekbouw van cDNA cruciaal, zelfs in het post-genoom rangschikkende Tijdperk. Hoewel cDNA het klonen van genen van belang door PCR een vereenvoudigde alternatieve route heeft verstrekt om transcriptstructuren zonder cDNA-bibliotheekbouw te analyseren, is de bouw van een cDNA-bibliotheek de benadering van keus wanneer een groot aantal cDNAs van één enkele mRNA-bron moet worden geanalyseerd. Tot op heden is er veel moeite gedaan om een methode te ontwikkelen voor de voorbereiding van hoogwaardige cDNA-bibliotheken (1-4). In tegenstelling, is de vraag hoe een hoogwaardige cDNA-bibliotheek van een kleine pool van RNAs voor te bereiden niet zo actief aangepakt. Nochtans, is dit een hoge prioriteit doel geworden omdat de onderzoekers vaak in hypothetische genen geà nteresseerd zijn die om slechts in bepaalde types van cellen of weefsels onder bepaalde voorwaarden worden voorspeld, zoals die in pathologische steekproeven worden gezien.
Er zijn een aantal rapporten die het gebruik van kleine hoeveelheden bronrna beschrijven voor het genereren van geamplificeerde cDNA of cRNA, waaruit targets voor microarray analyse kunnen worden voorbereid (5-15). Hun uiteindelijke doel is om volledig, niet-populatie-bevooroordeeld RNA te verkrijgen dat hoogst representatief is voor originele mRNA. Om dit uit te voeren, keuren deze methodes gewoonlijk PCR of transcriptie in vitro door de polymerase van T7 RNA goed om originele mRNA in de vorm van cDNA of cRNA, respectievelijk te versterken. Hoewel de vergelijking van uitdrukkingsprofielen één van de meest efficiënte benaderingen is om verschillen in de fysiologische Staten van cellen of weefsels te onderzoeken, kan de structurele karakterisering van afschriften (b.v., identificatie van alternatieve het verbinden patronen, de plaats van het transcriptiebegin, en de plaats van de transcriptiebeëindiging) niet door dergelijke microarray analyse worden gedaan. Sequencing analyse van elk transcript is onvermijdelijk in deze gevallen.
We ontwikkelden een methode die het mogelijk maakt een kleine hoeveelheid beginnend RNA te gebruiken om een cDNA bibliotheek te construeren die geschikt is voor het klonen van genen en uitgebreide sequencing analyse. De methode markeert de 3 ‘en 5’ einden van de eerste-ronde cDNAs met de opeenvolgingen van de phagepromotor van T7 en SP6, respectievelijk om Grootte-bias gevolgen tijdens de versterking te minimaliseren. Na twee-ronde Crna-versterking, zetten wij de versterkte cRNAs in cDNAs om, kloonden deze producten in plasmide, en evalueerden dan de resulterende cDNA-bibliotheek in vergelijking met die die door een conventionele methode wordt geconstrueerd (4,16,17).
materialen en methoden
cRNA-amplificatie
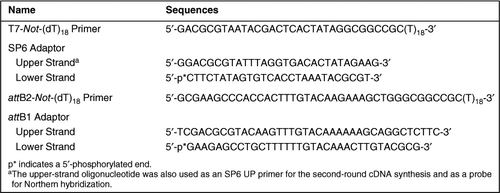
De in dit onderzoek gebruikte sequenties van oligonucleotiden zijn weergegeven in Tabel 1. Om het protocol vast te stellen, gebruikten we 1 µg totaal RNA bereid uit ICR muizenhersenen (8 weken oude mannelijke muizen) als sjabloon. Een mengsel (10 µL) van 1 µg totaal RNA en 100 pmol T7-Not-(dT)18 primer werd geïncubeerd bij 70°C gedurende 10 minuten en vervolgens snap-gekoeld op ijs. Dubbelstrengs cDNA synthese en adapter ligatie werden uitgevoerd volgens de protocollen van het superscript ® plasmide systeem (Invitrogen, Carlsbad, CA, USA) (4,16,17), met kleine wijzigingen. Kort, voor de eerste-streng cDNA synthese, 4 µL 5× eerste-streng buffer (Invitrogen), 1 µL 0,1 M dithiothreitol (DTT), 1 µL 10 mM dNTPs, 1 µL (40 U) RNaseOUT™ (Invitrogen), en 1 µL water werden toegevoegd aan de gedenatureerde RNA/T7-Not-(dT) 18 primer mixure en geïncubeerd bij 37°C gedurende 3 minuten om primers te ontharden. Dan, werden 2 µL (400 U) SUPERSCRIPT III RNase H-reverse transcriptase (Invitrogen) toegevoegd aan het reactiemengsel, en de temperatuur werd aangepast aan 50°C om de eerste bundel cDNA synthese te beginnen. Na 1 uur, de tweede-strand gen synthese werd uitgevoerd zoals hiervoor is beschreven door Gubler en Hoffman (18); de eerste-strand gen mengsel, 91 µL water, 30 µL 5× tweede deel buffer (Invitrogen), 3 µL 10 mM dNTPs, 1 µL (10 U) Escherichia coli DNA ligase, 4 µL (40 U) E. coli DNA-polymerase, en 1 µL (2 U) E. coli RNase H werden toegevoegd, en het mengsel werd geïncubeerd bij 16°C gedurende 2 uur. gen termini waren dan eind-gepolijst met 10 U T4-DNA-polymerase (Invitrogen) bij 16°C gedurende 5 min, en de reactie werd gestopt door de toevoeging van 10 ĩl 0,5 M ethyleendiamine-tetra-tetraacetic zuur (EDTA). De resulterende cDNA ‘ s werden gezuiverd door extractie met fenol/chloroform/isoamylalcohol (25:24:1), gevolgd door ethanolprecipitatie zoals eerder beschreven (17), met uitzondering van het gebruik van 1 µg gist tRNA in plaats van polyadenylzuur als drager. De neergeslagen cDNA werd opgelost in 50 µL Te (10 mM Tris-HCl, pH 8,0 en 1 mM EDTA), gemengd met 30 µL van een 20% (g/v) polyethyleenglycol (PEG) 6000/2, 5 M NaCl-oplossing, en vervolgens geïncubeerd op ijs gedurende 1 uur (17). Na de incubatie werd het cDNA-mengsel gedurende 15 minuten gecentrifugeerd bij 18.000× g bij 4°C. De resulterende cDNA-pellet werd tweemaal gespoeld met 70% ethanol, gedroogd en vervolgens geresuspendeerd in 30 µL water. De gezuiverde cDNAs werden ligated met 500 pmol SP6 adapter (Tabel 1) gebruikend 5 u T4 DNA ligase (Invitrogen) in een 50-µL reactievolume bij 16°C ‘ s nachts. Het adaptor-ligated mengsel werd tweemaal verdund met water, en vervolgens cDNAs werden gezuiverd met behulp van een dnaclear™ kolom (Ambion, Austin, TX, USA). Het eluaat (in 16 µL water) werd gebruikt als sjabloon voor cRNA-synthese. Met behulp van de Megascript ® T7 kit (Ambion) werden cRNAs ‘ s nachts gesynthetiseerd bij 37°C in een reactievolume van 40 µL. Nadat template cDNAs werden afgebroken door 4 U DNase I, werden de gesynthetiseerde cRNAs gezuiverd met behulp van een Rneasy® Mini Kit (QIAGEN, Valencia, CA, USA) en geëlueerd met 100 µL water. De concentratie van de cRNA werd bepaald door ultraviolet (UV) absorptie. Voor de tweede-ronde Crna versterking, 2 µg van de gesynthetiseerde cRNA en 100 pmol SP6 up primer (gelijk aan de bovenste-streng oligonucleotide van SP6 adapter getoond in Tabel 1) werden gebruikt als template en primer, respectievelijk. De tweede-ronde cDNA synthese werd uitgevoerd zoals in de originele omgekeerde transcriptie van bronrna, behalve dat SP6 omhoog primer het ontharden bij 50°C 3 min werd uitgevoerd. Na zuivering van verkregen double-stranded cDNA door een kolom dnaclear, werd 0.5 µg van cDNA gebruikt als malplaatje voor de volgende SP6 polymerase-bijgestane Crna versterking van RNA. Met behulp van de megascript SP6 kit (Ambion), cRNA synthese werd uitgevoerd bij 37°C gedurende 6 uur. nadat template cDNA werd gedegradeerd met behulp van DNase I, werden de gesynthetiseerde cRNAs gezuiverd zoals hierboven beschreven.
cDNA-Bibliotheekconstructie
twee microgram van de resulterende Crna ‘ s werden onderworpen aan dubbelstrengs-cDNA-synthese met 100 pmol attB2-Not – (DT)18 primer (Tabel 1). De cDNA-synthese, eind-afblunting, zuivering, en adapter-afbinding stappen werden uitgevoerd zoals in de vorige cDNA-syntheses, behalve dat de attB1-adapter (Tabel 1, 500 pmol) aan dubbelstrengs cDNAs werd geligeerd. Nadat de attB1-adapter-geligeerde cDNA ‘ s werden gezuiverd door opeenvolgende behandelingen van fenol/chloroform/isoamylalcohol-extractie, ethanolprecipitatie en PEG/NaCl-precipitatie in deze volgorde, werden ze opgelost in 50 µL TE en behandeld met RNase A (bij een uiteindelijke concentratie van 10 µg/mL; Invitrogen) om besmet RNA bij 37°C gedurende 30 min te degraderen. Het reactiemengsel werd opnieuw gezuiverd door extractie met fenol / chloroform/isoamylalcohol, gevolgd door ethanolprecipitatie en PEG / NaCl-precipitatie. De resulterende cDNAs werden opgelost in 15 µL TE, en hun hoeveelheid werd geschat op basis van fluorescente kleuring intensiteit na elektroforese op agarose gel (4). De attB1-ligated cDNAs (36 ng) werden onderworpen aan een in vitro recombinatiereactie (Gateway®-systeem; Invitrogen) met 250 ng attP-pSP73 donorvector zoals eerder beschreven (4,16,17). Kortom, de attB1-ligated cDNA en attP-pSP73 donor vector werden gemengd en geïncubeerd in een 10-µL reactievolume met 2 µL 5× BP Clonase™ reactiebuffer en 2 µL BP Clonase (beide van Invitrogen) bij 25°C ‘ s nachts. Het cDNA-mengsel werd gedurende 10 minuten behandeld met 2 µg proteïnase K (Invitrogen) bij 37°C om de reactie te blussen en geëxtraheerd met fenol/chloroform/isoamylalcohol, gevolgd door ethanolprecipitatie met 2 µg gist tRNA als drager. Het neergeslagen cDNA werd opgelost in 10 µL water en 1 µL van het mengsel werd gebruikt voor de transformatie van ElectroMAX™ DH10B™ E. coli cellen (Invitrogen). Na titratie van de resulterende cDNA-bibliotheek, werden de cDNA-plasmiden teruggevonden uit ongeveer 1.000.000 kolonies door een alkalische natriumdodecylsulfaat (SDS) methode zoals eerder beschreven (4,19). De resulterende cDNA klonen waren in een vorm van plasmide dragende attL plaatsen. Omdat de plasmiden die attB-plaatsen dragen in sommige toepassingen vereist zijn, werden deze plasmiden omgezet in die die attB-plaatsen dragen zoals eerder beschreven (16). Deze cDNA bibliotheek werd bijgevolg aangewezen als MB-AL (muis hersenen versterkte cRNA-afgeleide bibliotheek).
op dezelfde manier als beschreven voor MB-AL, construeerden we een cDNA-bibliotheek uit niet-amplified RNA (100 µg mouse brain total RNA) en noemden het MB-CL (mouse brain conventional constructed library).
Onderzoek van de Grootte-Bias Effect op de Versterkte cRNAs
om Te onderzoeken of de grootte-bias effect op cRNAs, de eerste ronde cRNAs getranscribeerd door T7-RNA-polymerase en de tweede-ronde-cRNAs getranscribeerd door SP6 RNA-polymerase werden uitgevoerd op 1,0% agarosegel (1 µg) en dan wiste op nylon membraan (Biodyne® B Nylon Membraan; PALL, East Hills, NY, USA). Ze werden gehybridiseerd met 32P-gelabelde SP6 up primer(Tabel 1) en niet-(dT)18 (5′-GCGGCCGCTTTTTTTTTTTTTTTT-3′) oligonucleotide, respectievelijk. Tien picomolen van elke sonde werden gelabeld met ATP (ongeveer 3000 Ci / mmol; Amersham Biosciences, Piscataway, NJ, USA) met behulp van T4 polynucleotide kinase (Takara, Kyoto Japan). Na ‘ s nachts hybridisatie in GMC-buffer (20) bij 45°C, werden deze membranen uitgebreid gewassen in 1× standaard zoutcitraat (SSC)/1% SDS-oplossing driemaal bij kamertemperatuur. Analyse van gehybridiseerde signalen werd uitgevoerd op een bas 2000 Image Analyzer (Fuji Photo Film, Tokyo, Japan)
evaluatie van cDNA-bibliotheken
gelelektroforese. Distributie van cDNA-insertgroottes werd onderzocht door elektroforese van cRNAs gesynthetiseerd door T7 RNA-polymerase met behulp van NotI-verteerd MB-AL en MB-CL plasmide cDNAs als sjablonen. Na zuivering met behulp van een RNeasy Mini Kit, werden de cRNAs elektroforesed op een formaldehyde-bevattende agarose gel met perfecte RNA Markers (EMD Biosciences, Madison, WI, USA). Na het kleuren van de gel met SYBR® Green II (Invitrogen), werd de fluorescerende kleuring intensiteit geanalyseerd met behulp van ImageQuant® (versie 5.0) Software op een FluorImager 595 (Amersham Biosciences).
willekeurige single-pass sequencing analyse. 3 ‘ – Eindopeenvolgingen van cDNA-klonen werden onderzocht door het rangschikken van DNA. Plasmide-DNA van 768 willekeurig gekozen klonen werden gezuiverd met behulp van een MFX-9600 Magnia® (Toyobo, Osaka, Japan) en geanalyseerd met een RISA 384-capillaire Sequencer (Shimadzu, Kyoto, Japan) (16). Na het trimmen van de vectorsequentie met behulp van Sequencher™ Software Versie 4.1 (Hitachi Software, Tokyo, Japan), werden de verkregen cDNA-sequenties die langer waren dan 200 nucleotide-residuen geclusterd met cDNA-vermeldingen in de GenBank® – database en onze interne muizendatabase. Het onderzoek naar de integriteit van de 3′ uiteinden van cDNA-klonen werd uitgevoerd door een analyse of er al dan niet een canonieke polyadenylatiesignaal hexameer (5′-AATAAA-3’) of de single-base varianten daarvan (11 signaalhexameren) werden gevonden binnen de 50 nucleotideresiduen stroomopwaarts van de poly(A) staart van cDNAs (21).
cDNA microarray. De microarray analyses werden uitgevoerd om cDNA populaties in de Mb-AL en MB-CL bibliotheken te vergelijken. Zelfgemaakte cDNA nylon microarrays met 3534 sondes werden voorbereid, en radio-isotopische detectie werd uitgevoerd (22). In het kort werden cDNA-plasmiden, die in ons instituut werden geïsoleerd en waarvoor volledige sequenties of eindsequenties al bekend waren, Gespot met GeneTAC™ RA1 (Genomic Solutions, Ann Arbor, MI, USA) op BioDyne B nylon membraan en vervolgens gefixeerd volgens de instructies van de fabrikant. De lijst van cdnas die op deze microarray wordt geà mmobiliseerd is beschikbaar op verzoek van de auteurs. MB-AL-en MB-CL-doel-cDNA ‘ s werden bereid met behulp van omgekeerde transcriptie van elk van de 1 µg cRNA-monsters die werden gebruikt voor de analyse van de grootte van de insert, zoals hierboven beschreven. Deze doelstellingen werden gesynthetiseerd en gelabeld in de reactie van het mengsel met 1 microgram van iedere cRNA, 100 ng willekeurige hexamer, 1× eerste deel buffer, 10 mM DTT, elk 800 µM dATP, dTTP, en dGTP, 800 nM, dCTP, 5 µL dCTP (>2500 Ci/mmol; Amersham Biosciences), en 100 U SuperScript II RNase H – reverse transcriptase bij kamertemperatuur gedurende 10 minuten en daarna op 42°C gedurende 1 uur. Na alkaline lysis en neutralisatie, de gelabeld cDNAs werden gezuiverd met de QIAquick® PCR zuivering kit (QIAGEN) en geteld. Target cDNA werd onderworpen aan hybridisatie met de cDNA nylon microarray in aanwezigheid van 1 µg polyadenylzuur en 2,5 µg muis Cot-1 DNA® (Invitrogen) in 250 µL PerfectHyb™ buffer (Toyobo) bij 68°C ‘ s nachts. Na streng wassen bij 68°C met 2 × SSC / 1% SDS (twee wasbeurten van elk 15 min) en vervolgens met 0,1× SSC / 1% SDS (twee wasbeurten van elk 30 min), werden de hybridisatiesignalen gedetecteerd en geanalyseerd op een FLA-8000 (Fuji fotofilm). De cDNA-vlekken die sterkere signalen tonen dan die van de achtergrondcontrole werden geselecteerd en onderworpen aan verdere analyse. Na het uitvoeren van globale normalisatie, werden scatter plots van signaalintensiteiten van cDNA spots afkomstig van MB-AL en MB-CL targets verkregen.
RNA blot hybridisatie. De blotkruising van RNA werd uitgevoerd om Grootte-bias gevolgen te onderzoeken. De Mb-AL en MB-CL cRNAs (elk 1,5 µg, gesynthetiseerd zoals hierboven beschreven) werden elektroforesed op formaldehyde-bevattende agarose gel en overgebracht naar BioDyne B nylon membraan. De sonde cDNAs werden voorbereid gebruikend PCR-versterking. Primers werden ontworpen op basis van de sequenties geregistreerd in de GenBank database of onze in-house database; sjablonen waren de cDNA plasmiden die we hadden geïsoleerd. De sonde van ribosomaal S6-eiwit was 724 bp in lengte, versterkt met 5′-CGCTCGGCTGTGTCAAGATG-3′ en 5′-gaggacagcctacgtctctggg-3′ primers, en de sonde van heat shock protein (hsp) 70, 940 BP in lengte, werd versterkt met 5′-GATGGACAAGGCGCAGATCC-3′ en 5′-CTCGATGGGGGTCCTGAGC-3′ primers. Deze sondes werden geëtiketteerd met dCTP (ongeveer 3000 Ci/mmol; Amersham Biosciences) gebruikend het RadPrime DNA-etiketteringssysteem (Invitrogen). Na hybridisatie ‘ s nachts in PerfectHyb buffer bij 65°C, werden de membranen achtereenvolgens gewassen met 0,1× SSC/1% SDS bij kamertemperatuur gedurende 5 min en gedurende 15 min, en vervolgens bij 65°C gedurende 30 min. De hybridisatiesignalen werden ontdekt op een bas 2000 beeldanalysator.
resultaten en discussie
De cDNA-bibliotheekconstructie die we in deze studie hebben ontwikkeld, is weergegeven in Figuur 1 (nieuw geïntroduceerde stappen worden gemarkeerd met een ster). Deze methode bestaat uit twee rondes van cRNA-versterkingsstap, omzetting van cRNAs in cDNAs, en recombinational het klonen in een plasmide. De belangrijkste stap in deze methode is de adapterafbinding aan markering cDNA eindigt met SP6 promotoropeenvolging. Zodra wij cDNAs in dit formaat met succes kunnen samenstellen, wordt het mogelijk om de eerste-ronde cRNAs specifiek om te keren die de SP6 promotoropeenvolging bij hun 3′ eind bevatten. Second-round cRNAs wordt samengesteld met de polymerase van SP6 RNA gebruikend resulterende double-stranded cDNAs als malplaatjes. Na de twee rondes van polymerase-bijgestane versterking van RNA, worden de resulterende cRNAs dan omgezet in double-stranded cDNAs door omgekeerde transcriptie gebruikend attB2-niet-(dT)18 inleiding. Het gebruik van attB2-niet-(dT)18 primer in de omgekeerde transcriptie laat ons toe om slechts cRNAs om te zetten die de opeenvolging 5′-(a)18GCGGCCGC-3′ aan hun 3′ einden dragen. Omdat deze wijzigingen slechts versterking en het klonen van cDNAs zouden moeten toestaan die einden correct geëtiketteerd hebben, verwachten wij dat deze methode groottebias tijdens de polymerase-bijgestane versterking van RNA zal minimaliseren.

de stappen in de adapter-ligatie-ondersteunde cRNA-versterking en de volgende cDNA-bibliotheekconstructie worden geïllustreerd. Om de versterkte cDNA-grootte met die van eerste-ronde cDNAs te handhaven, introduceerden wij sommige wijzigingen in conventionele polymerase-bijgestane methodes van RNA (die met sterren worden benadrukt). De blauwe en rode lijnen wijzen op cDNA en RNA, respectievelijk. E. coli, Escherichia coli.
Om het testen van de werkzaamheid van de methode, die we gebouwd gen bibliotheken door een conventionele methode (16) met behulp van nonamplified totaal RNA van muis hersenen (100 µg) en door de hierboven beschreven methode met 1 µg totaal RNA van muis hersenen. Tabel 2 geeft een overzicht van de hoeveelheden cDNA en cRNA verkregen in elke stap, berekend als een gemiddelde van drie onafhankelijke experimentele runs van MB-AL constructie. We behaalden een gemiddelde van 6,4×107 onafhankelijke cDNA klonen als de uiteindelijke output. Omdat dit aantal cDNA-klonen afkomstig is van een totaal van 36 ng cDNA gegenereerd met behulp van het amplificatieprotocol, berekenen we dat deze methode ongeveer 1,2×1011 cDNA-klonen oplevert uit 1 µg totaal RNA.

om het size-bias effect tijdens amplificatiestappen experimenteel te onderzoeken, vergeleken we vervolgens de groottedistributies van de eerste-ronde en de tweede-ronde cRNA door RNA blot hybridisatie analyse. In deze experimenten, werden de eerste-ronde en de tweede-ronde cRNAs gekruist met SP6 omhoog primer en met niet-(DT)18 oligonucleotide, respectievelijk, omdat deze oligonucleotidesondes slechts cRNAs correct aan het eind ontdekken getranscribeerd. Zoals getoond in Figuur 2, werd de grootte van RNA bij de piek van kruissignaal in de tweede-ronde cRNA gevonden om bijna hetzelfde te zijn als dat in de eerste-ronde cRNA, maar met een lichte verschuiving naar kleinere grootte. We vermoeden dat het afkappen van Crna ‘ s waarschijnlijk plaatsvond tijdens de tweede ronde cDNA synthese met de SP6 UP primer.
de blothybridisatie van RNA werd uitgevoerd om het effect van grootte-bias op de cRNAs te onderzoeken. De kruissignalen langs de richting van cRNA-elektroforese werden uitgezet. De RNA-grootte werd bepaald op basis van de mobiliteit van de RNA-laddermarker. Blauwe lijn, de eerste ronde cRNA gehybridiseerd met SP6 up probe; rode lijn, de tweede ronde cRNA gehybridiseerd met niet – (dT)18 probe; PSL-waarde, signaalintensiteit willekeurig aangegeven door de Image Gauge Software (Fuji Photo Film).
om de kwaliteit van de resulterende versterkte cDNA-bibliotheek verder te beoordelen, hebben we ook verschillende functies vergeleken van de cDNA-bibliotheken gegenereerd met en zonder de versterking (respectievelijk MB-AL en MB-CL). We onderzochten eerst de grootteverdeling van de cDNA-wisselplaten, zoals getoond in Figuur 3A. de resultaten van de signalen van de fluorescentieintensiteit verkregen van het gel-kleurende beeld gaven aan dat de cDNA-wisselplaatgroottes van MB-AL en MB-CL gelijkaardig waren, maar het piekpunt was iets kleiner in MB-AL (0,65 kb) vergeleken met dat in MB-CL (0,8 kb). Vervolgens analyseerden we 3 ‘ – end sequenties van willekeurig gesamplede cDNA klonen uit elke bibliotheek. De clustering resultaten voor deze expressed sequence tags (ESTs) zijn weergegeven in Figuur 3B en suggereren dat de complexiteit van MB-AL even hoog was als die van MB-CL. Echter, het is vermeldenswaard dat zeer redundante cDNA klonen verdwenen in MB-AL. Door een statistische analyse (Chi-test) werd aangetoond dat het verschil in gedwongen ontslagen tussen MB-AL en MB-CL significant was (de kans dat de gedwongen ontslagen In MB-AL en MB-CL vergelijkbaar zijn is <0,001). Bovendien, onderzochten wij de integriteit van 3 ‘ einden van cDNAs door te analyseren of elk EST een polyadenylation signaal hexamer of niet bevatte. Uit de resultaten bleek dat 80,1% en 87,3% van de cDNA-klonen in respectievelijk MB-AL en MB-CL plausibele polyadenylatiesignaalsequenties bevatten (21). De afname van de incidentie van polyadenylatie signaal hexameren in MB-AL wees waarschijnlijk op een toename van het aantal cDNA klonen waar cDNA synthese intern geprimed werd als gevolg van twee rondes van DT priming.

MB-AL (mouse brain amplified cRNA-derived library) werd geëvalueerd in vergelijking met een conventioneel geconstrueerde library, MB-CL. (A) vergelijking van de cDNA-lengtes van de insert. In vitro getranscribeerde Crna ‘ s werden gefractioneerd op agarose gel en gekleurd. Hun fluorescente intensiteiten langs de richting van elektroforese (aangegeven als RNA-grootte) worden getoond in relatieve fluorescente eenheden (RFU ‘ s). B) Clustering van de resultaten van MB-AL en MB-CL 3’-eindsequenties (ESTs). (C) vergelijking van de cDNA-populatie in MB-AL en MB-CL. De signalen die door microarray kruising worden verkregen werden uitgezet. (D) RNA blot hybridisatie resultaten over ribosomal S6 eiwit en HSP 70. MB-al cRNAs waren in de linker rijstrook en die van MB-CL waren in de rechter rijstrook (aangegeven door respectievelijk AL en CL). Pijlpunten wijzen op de posities van de voorspelde RNA-grootte van elk gen. hsp, hitteschok proteïne.
door microarray hybridisatie hebben we de cDNA populatie van MB-AL vergeleken met die van MB-CL. Figuur 3C toont een spreidingsgrafiek van hybridisatiesignalen van MB-AL en MB-CL doelen. De resultaten wezen op een aanzienlijk hoge correlatie van hybridisatiesignalen tussen Mb-AL en MB-CL doelen, die suggereren dat er geen schadelijk effect van de versterking in cDNA populatie was.
ten slotte hebben we RNA blot hybridisatie analyse uitgevoerd om de grootte-bias effecten te onderzoeken met aandacht voor huishoudgenen (figuur 3D). Voor ribosomaal S6-eiwit en hsp 70 waren de cRNA-groottes in zowel MB-AL als MB-CL vergelijkbaar en vergelijkbaar met die welke verwacht werden van hun gerapporteerde nucleotidesequenties. Het hybridisatiepatroon van HSP 70 cRNAs in MB-AL verschilde echter enigszins van dat in MB-CL, wat erop wijst dat het effect van groottebias niet volledig werd verwijderd.
de methode van cDNA-bibliotheekconstructie die in dit rapport wordt beschreven, werd ontworpen om groottevooroordelen tijdens amplificatiestappen te minimaliseren die door RNA-polymerase worden bijgestaan. Dergelijke de polymerase-bijgestane methode van de bibliotheekbouw van RNA van een kleine pool van RNA voor uitvoerige het rangschikken analyse is niet goed bestudeerd, terwijl cDNA/cRNA de methodes van de bibliotheekbouw voor microarray kruising actief zijn nagestreefd. Hoewel Lukyanov et al. (23) en Piao et al. (24) gerapporteerde methoden die PCR-versterking gebruiken om een cDNA-bibliotheek van een submicrogram-hoeveelheid totaal RNA te construeren, heeft PCR-versterking intrinsiek nadelen zoals strenge grootte en populatievooroordeel en lage trouw van cDNA-versterking. Dit was de reden waarom wij in deze studie een cDNA-bibliotheekbouwmethode probeerden te ontwikkelen die op polymerase-bijgestane versterking van RNA wordt gebaseerd.
hiervoor hebben we een nieuwe RNA-polymerase-ondersteunde amplificatiestrategie ontwikkeld, zoals weergegeven in Figuur 1, omdat de methoden van Eberwine et al. (5), Huang et al. (10), en Lin et al. (11) nog steeds nadelen voor de bereiding van een plasmide-bevattende cDNA geschikt voor uitgebreide genstructuur analyse. In de eerste, cDNA synthese na Crna versterking wordt gedaan met willekeurige hexamers, en dit maakt de versterkte cDNAs onvermijdelijk korter dan de originele degenen (5,7). In de laatste, is de introductie van een homopolymere staart aan het eerste-streng cDNA-eind door einddeoxynucleotidyltransferase (TdT) of Moloney murine leukemia virus (MMLV) omgekeerde transcriptase activiteit (10,11) zinvol om het grootte-bias effect tijdens de polymerase-geassisteerde versterking van RNA zoals in onze methode te minimaliseren. Nochtans, is de homopolymere tailing methode gekend om truncation van cDNAs toe te schrijven aan onverwacht priming van cDNA synthese van een homopolymere strook van interne opeenvolgingen van RNA te veroorzaken. Omdat de adapter-ligatiemethode die in deze studie wordt beschreven een specifieke opeenvolging voor cDNA-priming kon verstrekken, konden wij verwachten om de omvang van cDNA-afkaping gebruikend onze methode te verminderen.
in principe zou onze methode alleen de cDNA ‘ s moeten versterken geflankeerd door een intrinsieke Poly(a) staart en de SP6 adaptersequentie, die werden gegenereerd in de eerste ronde cDNA synthese. De resultaten in Figuur 2 en figuur 3A waren in wezen consistent met dit, hoewel de cDNA-populatie in de kleinere grootte range was een beetje toegenomen. De resultaten van RNA blot analyse (figuur 3D) toonden ook aan dat de grootte-bias effect kan worden verminderd, maar niet volledig verwijderd, zelfs in onze methode. Dit was waarschijnlijk omdat afgekapt cDNAs toegevoegd door een poly (a) staart en de SP6 promotoropeenvolging tijdens omzetting van cRNA in cDNA werden gegenereerd. Deze mening werd ondersteund door het feit dat de frequentie van het optreden van de polyadenylatiesignaalsequenties in MB-AL significant lager was dan die waargenomen in de conventionele cDNA-bibliotheek (21). Afkaping van cDNAs kan optreden tijdens cDNA synthese als gevolg van interne priming van RNA met de DT-tailed primer en de SP6 primer. Omdat homopolymeerrekkingen vaak voorkomen in eukaryotic mRNAs, kan de homopolymeer tailing methode strengere groottevooringenomenheid opleggen op multiple-round Crna versterking dan onze adapter-ligation methode. Hoewel het verhogen van de reactietemperatuur tijdens omgekeerde transcriptie en het verminderen van de inleidingsconcentratie het afwijkende interne priming in cDNA-synthese tot op zekere hoogte zou kunnen onderdrukken, is het gekend om moeilijk te zijn om dit op dit moment volledig te elimineren. Aldus, hoewel wij twee-ronde Crna versterking voor demonstratie van het algemene protocol hebben uitgevoerd, in de praktijk, adviseren wij het overslaan van de tweede-ronde Crna versterking wanneer genoeg cRNA in de eerste-ronde reactie kan worden verkregen.
rekening houdend met deze resultaten, concluderen we dat onze nieuwe methode nuttig is voor de opbouw van cDNA-bibliotheken vanuit een kleine hoeveelheid beginnend RNA. In feite, hebben wij met succes en routinematig deze methode voor de bouw van cDNA-bibliotheken gebruikt wanneer de hoeveelheid totaal RNA minder dan 1 µg is (getoonde gegevens niet). Omdat 1 µg totaal RNA van 105-106 zoogdiercellen of 1 mg zoogdierweefsels zou kunnen worden teruggekregen, kunnen cDNA-bibliotheken gemakkelijk van cellen worden geconstrueerd die door celsorteerders of microdissection door onze methode te gebruiken worden verdeeld. Bovendien, omdat we berekenen dat deze methode ons in staat stelt om meer dan 105 cDNA klonen te verkrijgen van 1 pg totaal RNA, wat dicht bij de hoeveelheid totaal RNA in een enkele cel ligt, zou een enkele cel-afgeleide cDNA bibliotheek kunnen worden geconstrueerd op basis van deze adapter-ligatie-ondersteunde cRNA amplificatie.de auteurs zijn dankbaar voor de instructie van statistische analyse van Dr.Kazuharu Misawa; verdeling van plasmide DNA ‘ s van Dr. Hisashi Koga; en technische hulp van mevrouw Akiko Ukigai-Ando en de Heer Takashi Watanabe. De studie werd ondersteund door een subsidie van het Kazusa DNA Research Institute aan R. O., R. F. K. en O. O.
concurrerende belangenverklaring
De auteurs verklaren geen concurrerende belangen.
- 1. Carninci, P., C. Kvam, A. Kitamura, T. Ohsumi, Y. Okazaki, M. Itoh, M. Kamiya, K. Shibata, et al.. 1996. Hoge efficiëntie cDNA klonen door biotinylated cap trapper. Genomics 37: 327-336.Crossref, Medline, CAS, Google Scholar
- 2. Ohara, O., T. Nagase, K. Ishikawa, D. Nakajima, M. Ohira, N. Seki, en N. Nomura. 1997. Bouw en karakterisering van menselijke hersenen cDNA bibliotheken geschikt voor analyse van cDNA klonen coderen relatief grote eiwitten. DNA res. 4: 53-59.Crossref, Medline, CAS, Google Scholar
- 3. Suzuki, Y., K. Yoshitomo-Nakagawa, K. Maruyama, A. Suyama en S. Sugano. 1997. Bouw en karakterisering van een full length-verrijkte en een 5′-end-verrijkte cDNA bibliotheek. Gene 200: 1149-1156.Crossref, Google Scholar
- 4. Ohara, O. en G. Temple. 2001. Directionele cDNA-bibliotheekbouw die door de in vitro recombinatiereactie wordt bijgestaan. Nucleïnezuren res. 29: e22.Crossref, Medline, CAS, Google Scholar
- 5. Eberwine, J., H. Yeh, K. Miyashiro, Y. Cao, S. Nair, R. Finnel, M. Zettel, en P. Coleman. 1992. Analyse van genexpressie in enkele levende neuronen. Proc. Natl. Acad. Sci. USA 89: 3010-3014.Crossref, Medline, CAS, Google Scholar
- 6. Wang, E., L. D. Miller, G. A. Ohnmacht, E. T. Liu, and F. M. Marincola. 2000. High-fidelity mRNA versterking voor Gen profilering. Nat. Biotechnol. 18:457–459.Crossref, Medline, CAS, Google Scholar
- 7. Baugh, L. R., A. A. Hill, E. L. Brown en C. P. Hunter. 2001. Kwantitatieve analyse van mRNA amplificatie door in vitro transcriptie. Nucleïnezuren res. 29: e29.Crossref, Medline, CAS, Google Scholar
- 8. Zhumabayeva, B., L. Diatchenko, A. Chenchik, and P. D. Siebert. 2001. Gebruik van SMART™ – gegenereerde cDNA voor genexpressiestudies in veelvoudige menselijke tumors. BioTechniques 30: 158-163.Link, CAS, Google Scholar
- 9. Gomes, L., R. Silva, B. S. Stolf, E. B. Cristo, R. Hirata, Jr., F. A. Soares, L. Reis, E. J. Neves, et al.. 2003. Vergelijkende analyse van versterkt en niet-amplified RNA voor hybridisatie in cDNA microarray. Anaal. Biochem. 321:244–251.Crossref, Medline, CAS, Google Scholar
- 10. Huang, J., T. Lin, D. Chang, S. Lin, en S. Ying. 2003. Afgekapt Bcl-2, een potentiële pre-metastische marker bij prostaatkanker. Biochem. Biophys. Res. Commun. 306:912–917.Crossref, Medline, CAS, Google Scholar
- 11. Lin, S. L. En S. Y. Ying. 2003. mRNA / cDNA-bibliotheekbouw gebruikend RNA-polymerase het cirkelen reactie. Methoden Mol. Biol. 221:129–143.Medline, CAS, Google Scholar
- 12. Matz, M. V. 2003. Versterking van representatieve cDNA-pools uit microscopische hoeveelheden dierlijk weefsel. Methoden Mol. Biol. 221:103–116.Medline, CAS, Google Scholar
- 13. Petalidis, L., S. Bhattacharyya, G. A. Morris, V. P. Collins, T. C. Freeman, and P. A. Lyons. 2003. Globale versterking van mRNA door template-switching PCR: lineariteit en toepassing op microarray analyse. Nucleïnezuren res. 22: e142.Crossref, Google Scholar
- 14. Polacek, D. C., A. G. Passerini, C. Shi, N. M. Francesco, E. Manduchi, G. R. Grant, S. Powell, H. Bischof, et al.. 2003. Getrouwheid en verhoogde gevoeligheid van differentiële transcriptieprofielen na lineaire versterking van nanogramhoeveelheden endothelial mRNA. Fysiol. Genomics 13: 147-156.Crossref, Medline, CAS, Google Scholar
- 15. Wilson, C., S. D. Pepper, Y. Hey, en C. J. Miller. 2004. De versterkingsprotocollen introduceren systematische maar reproduceerbare fouten in de studies van de genuitdrukking. BioTechniques 36: 498-506.Link, CAS, Google Scholar
- 16. Ohara, O., T. Nagase, G. Mitsui, H. Kohga, R. Kikuno, S. Hiraoka, Y. Takahashi, S. Kitajima, et al.. 2002. Karakterisering van de grootte-gefractioneerde cDNA-bibliotheek die door de in vitro recombinatie-bijgestane methode wordt geproduceerd. DNA res. 9: 47-57.Crossref, Medline, CAS, Google Scholar
- 17. Ohara, O. 2003. Bouw van grootte-gefractioneerde cDNA-bibliotheek die door een in vitro recombinatiereactie wordt bijgestaan. Methoden Mol. Biol. 221:59–71.Medline, CAS, Google Scholar
- 18. Gubler, U. En B. J. Hoffman. 1983. Een eenvoudige en zeer efficiënte methode voor het genereren van cDNA bibliotheken. Gen 25: 263-269.Crossref, Medline, CAS, Google Scholar
- 19. Sambrook, J. en D. W. Russell. 2001. Bereiding van plasmide-DNA door alkalische lysis met SDS, blz. 1.31-1.42. Molecular Cloning, (3rd edition). CSH Laboratoriumpers, Cold Spring Harbor, NY.Google Scholar
- 20. Church, G. M., W. Gilbert. 1984. Genomische sequencing. Proc. Natl. Acad. Sci. USA 81: 1991-1995.Crossref, Medline, CAS, Google Scholar
- 21. Beaudoing, E., S. Freier, J. R. Wyatt, J. M. Claverie, en D. Gautheret. 2000. Patronen van variant polyadenylation signaalgebruik in menselijke genen. Genome Res. 10: 1001-1010.Crossref, Medline, CAS, Google Scholar
- 22. Tanaka, T. S., S. A. Jaradat, M. K. Lim, G. J. Kargul, X. Wang, M. J. Grahovac, S. Pantano, Y. Sano, et al.. 2000. Het genoom-brede uitdrukking profileren van mid-drachtatie placenta en embryo gebruikend een microarray van 15.000 muisontwikkeling cDNA. Proc. Natl. Acad. Sci. USA 97: 9127-9132.Crossref, Medline, Google Scholar
- 23. Lukyanov, K., L. Diatchenko, A. Chenchik, A. Nanisetti, P. Siebert, N. Usman, M. Matz, en S. Lukyanov. 1997. De bouw van cDNA-bibliotheken van kleine hoeveelheden totaal RNA gebruikend het effect van supressionpcr. Biochem. Biophys. Res. Commun. 230:285–288.Crossref, Medline, CAS, Google Scholar
- 24. Piao, Y., N. T. Ko, M. K. Lim, en M. Ko. 2001. De bouw van lang-afschriften verrijkte cDNA-bibliotheken van submicrogram-bedragen van totale RNAs door een universele PCR-versterkingsmethode. Genome Res. 11: 1553-1558.Crossref, Medline, CAS, Google Scholar
