Introduksjon
den komplette genomsekvenser av ulike organismer, inkludert pattedyr, har nylig blitt tilgjengelig som en konsekvens av raske fremskritt I DNA-sekvensering teknologi. Men spesielt hos pattedyr spiller analysen av transkripsjoner fortsatt en nøkkelrolle i å bygge bro over gapet mellom genomet og proteomet. Dette skyldes hovedsakelig at vi for tiden ikke nøyaktig kan forutsi strukturer av transkripsjoner avledet fra et bestemt gen fra den genomiske informasjonen alene. Således, som en metode for analyse av transkripsjoner, cDNA bibliotek konstruksjon er avgjørende, selv i post-genom sekvensering æra. Selv om cDNA kloning av gener av INTERESSE VED PCR har gitt en forenklet alternativ rute for å analysere transkripsjon strukturer uten cDNA bibliotek konstruksjon, er byggingen av et cDNA bibliotek tilnærming av valget når et stort antall cdna fra en enkelt mRNA kilde skal analyseres. Til dags dato har det blitt lagt stor vekt på å utvikle en metode for utarbeidelse av cDNA-biblioteker av høy kvalitet (1-4). I motsetning til dette er spørsmålet om hvordan å forberede et cDNA-bibliotek av høy kvalitet fra et lite basseng Av Rna ikke så aktivt adressert. Dette har imidlertid blitt et høyt prioritert mål fordi forskere ofte er interessert i hypotetiske gener som forventes å bli uttrykt bare i visse typer celler eller vev under spesielle forhold, som de som er sett i patologiske prøver.
Det har vært en rekke rapporter som beskriver bruken av små mengder kilde-RNA for å generere forsterket cDNA eller cRNA, hvorfra mål for mikroarray-analyse kan utarbeides (5-15). Deres endelige mål er å oppnå full lengde, ikke-populasjonsforspent RNA som er svært representativ for den opprinnelige mRNA. FOR å utføre dette, bruker disse metodene VANLIGVIS PCR eller in vitro transkripsjon Av T7 RNA-polymerase for å forsterke den opprinnelige mRNA i form av henholdsvis cDNA eller cRNA. Selv om sammenligningen av uttrykksprofiler er en av de mest effektive tilnærmingene for å undersøke forskjeller i de fysiologiske tilstandene til celler eller vev, kan ikke strukturell karakterisering av transkripsjoner (f. eks. identifisering av alternative spleisingsmønstre, transkripsjonsstartsted og transkripsjonsterminasjonssted) gjøres ved en slik mikroarray-analyse. Sekvenseringsanalyse av hvert transkripsjon er uunngåelig i disse tilfellene.
Vi utviklet en metode som tillater en liten mengde start RNA som skal brukes til å konstruere en cDNA bibliotek egnet for genet kloning og omfattende sekvensering analyse. Metoden koder 3′ og 5 ‘ ender av første runde cDNAs med Henholdsvis t7 og SP6 fagpromotor sekvenser for å minimere størrelsesbiaseffekter under forsterkningen. Etter to-runde cRNA-forsterkning konverterte vi de forsterkede crna til cdna, klonet disse produktene til et plasmid, og evaluerte deretter det resulterende cDNA-biblioteket i sammenligning med det som ble konstruert ved en konvensjonell metode (4,16,17).
Materialer og metoder
Crna Amplifikasjon
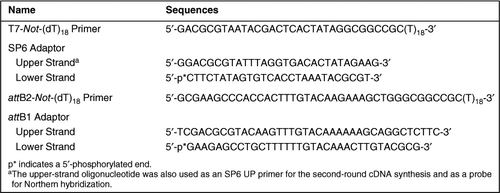
sekvensene av oligonukleotider brukt i denne studien er vist I Tabell 1. For å etablere protokollen brukte vi 1 µ totalt rna fremstilt FRA ICR musehjerne (8 uker gamle hannmus) som mal. En blanding(10 µ) av 1 µ total RNA og 100 pmol T7-Ikke-(dT)18 primer ble inkubert ved 70°C i 10 min og deretter hurtigkjølt på is. Dobbeltstrenget cDNA-syntese og adapterligering ble utført etter protokollene FRA HEVET plasmid-Systemet (Invitrogen, Carlsbad, CA, USA) (4,16,17), med mindre modifikasjoner. Kort, for det første-strand cDNA syntese, 4 µL 5× første-strand buffer (Invitrogen), 1 µL 0,1 M ditiotreitol (DTT), 1 µL 10 mM dntp, 1 µL (40 U) RNaseOUT™ (Invitrogen), og 1 µL vann ble lagt til denaturert RNA/T7-Ikke-(dT)18 primer mixure og inkubert ved 37°C i 3 min å bindes spesifikt primere. Deretter ble 2 µ (400 E) hevet III RNase H-revers transkriptase (Invitrogen) tilsatt til reaksjonsblandingen, og temperaturen ble justert til 50°C for å starte cDNA-syntesen med første tråd. Etter 1 t, den andre-strand cDNA syntese ble gjennomført som tidligere beskrevet av Gubler og Hoffman (18); for det første-strand cDNA blanding, 91 µL vann, 30 µL 5× andre-strand buffer (Invitrogen), 3 µL 10 mM dntp, 1 µL (10 U) Escherichia coli DNA ligase, 4 µL (40 U) E. coli DNA-polymerase, og 1 µL (2 U) E. coli RNase H er lagt til, og blandingen ble inkubert ved 16°C for 2 h. cDNA termini var da slutt-polert med 10 U T4 DNA-polymerase (Invitrogen) ved 16°C i 5 min, og reaksjonen ble stoppet ved tillegg av 10 µL 0,5 M etylendiamin tetraacetic syre (EDTA). De resulterende cdnaene ble renset ved ekstraksjon med fenol/kloroform / isoamylalkohol (25:24:1), etterfulgt av etanolutfelling som tidligere beskrevet (17), unntatt bruk av 1 µ gjær tRNA i stedet for polyadenylsyre som bærer. Den utfelte cDNA ble oppløst i 50 µ te (10 Mm Tris-HCl, pH 8,0 OG 1 mM EDTA), blandet med 30 µ av en 20% (w/v) polyetylenglykol (PEG) 6000/2, 5 M NaCl-oppløsning, og deretter inkubert på is i 1 time (17). Etter inkuberingen ble cDNA-blandingen sentrifugert ved 18 000× g ved 4°C i 15 min. Den resulterende cDNA-pelleten ble skyllet to ganger med 70% etanol, tørket og deretter resuspendert i 30 µ vann. De rensede cdnaene ble ligert med 500 pmol SP6-adapter (Tabell 1) ved bruk av 5 U T4 DNA-ligase (Invitrogen) i et 50-µ reaksjonsvolum ved 16°. Den adapterligerte blandingen ble fortynnet to ganger med vann, og deretter ble cdna renset ved Hjelp Av En DNAclear™ – kolonne (Ambion, Austin, TX, USA). Eluatet (i 16 µ vann) ble brukt som mal for cRNA-syntese. Ved Bruk Av Megascript® T7-settet (Ambion) ble crna syntetisert ved 37°C over natten i et 40-µ reaksjonsvolum. Etter at mal-cdnaer ble degradert av 4 U DNase I, ble de syntetiserte crnaene renset ved hjelp Av Et Rneasy® Minisett (QIAGEN, Valencia, CA, USA) og eluert med 100 µ vann. Konsentrasjonen av cRNA ble bestemt ved ultrafiolett (UV) absorpsjon. For den andre runde cRNA-forsterkningen ble 2 µ av den syntetiserte cRNA og 100 pmol SP6 UP-primeren (lik det øvre streng oligonukleotid AV SP6-adapteren vist i Tabell 1) brukt som henholdsvis mal og primer. CDNA-syntesen i andre runde ble utført som i den opprinnelige revers-transkripsjonen fra kilde-RNA, bortsett fra AT sp6 OPP-primerglødning ble utført ved 50°C i 3 min. Etter rensing av den oppnådde dobbeltstrengede cDNA gjennom En DNAclear-kolonne ble 0,5 µ av cDNA brukt som en mal for den NESTE SP6 rna-polymeraseassistert cRNA-forsterkning. Ved Hjelp Av Megascript SP6 kit (Ambion) ble cRNA-syntese utført ved 37°C i 6 timer. etter at cDNA-malen ble degradert ved Bruk Av DNase I, ble de syntetiserte crnaene renset som beskrevet ovenfor.
cDNA Bibliotekkonstruksjon
To mikrogram av de resulterende crnaene ble utsatt for dobbeltrådet cDNA-syntese ved bruk av 100 pmol attB2-Ikke-(dT)18 primer (Tabell 1). CDNA-syntese, end-blunting, rensing og adapter-ligeringstrinn ble utført som i de forrige cDNA-syntesene, bortsett fra at attB1-adapteren (Tabell 1500 pmol) ble ligert til de dobbeltstrengede cdnaene. Etter at attB1 adaptor-ligerte cdnaer ble renset ved suksessive behandlinger av fenol / kloroform / isoamylalkoholekstraksjon, etanolutfelling og PEG/NaCl-nedbør i denne rekkefølgen, ble de oppløst i 50 µ te og behandlet Med RNase A (ved en endelig konsentrasjon på 10 µ / mL; Invitrogen) for å nedbryte forurenset RNA ved 37°C i 30 min. Reaksjonsblandingen ble igjen renset ved ekstraksjon med fenol / kloroform / isoamylalkohol, etterfulgt av etanol utfelling og PEG / NaCl utfelling. De resulterende cdnaene ble oppløst i 15 µ TE, og deres mengde ble estimert fra fluorescerende fargeintensitet etter elektroforese på agarosegel (4). AttB1-ligerte cdnaer (36 ng) ble utsatt for en in vitro rekombinasjonsreaksjon (Gateway® System; Invitrogen) med 250 ng attP-pSP73 donorvektor som tidligere beskrevet (4,16,17). I korte trekk, den attB1-ligated cDNA og attP-pSP73 donor vektor ble blandet og inkubert i en 10-µL reaksjon volum med 2 µL 5× BP Clonase™ reaksjon buffer og 2 µL BP Clonase (både fra Invitrogen) ved 25°C over natten. CDNA-blandingen ble behandlet med 2 µ proteinase K (Invitrogen) ved 37°C i 10 minutter for å slukke reaksjonen og ekstrahert med fenol/kloroform/isoamylalkohol, etterfulgt av etanolutfelling med 2 µ gjær tRNA som bærer. Den utfelte cDNA ble oppløst i 10 µ vann, og 1 µ av blandingen ble brukt til transformasjon Av ElectroMAX™ DH10B™ E. coli celler (Invitrogen). Etter titrering av det resulterende cDNA-biblioteket ble cDNA-plasmidene gjenvunnet fra omtrent 1 000 000 kolonier ved hjelp av en alkalisk natriumdodecylsulfat (sds) – metode som tidligere beskrevet (4 19). De resulterende cDNA kloner var i en form for plasmid bærer attL områder. Fordi plasmider som bærer attB-steder, kreves i enkelte applikasjoner, ble disse plasmidene omdannet til de som bærer attB-steder som tidligere beskrevet (16). Dette cDNA-biblioteket ble følgelig betegnet SOM MB-AL (mouse brain amplified cRNA-derived library).
på samme måte som BESKREVET FOR MB-AL, konstruerte vi et cDNA-bibliotek fra nonamplified RNA (100@g mouse brain total RNA) og betegnet DET SOM MB-CL (mouse brain conventionally constructed library).
Undersøkelse Av Size-Bias-Effekt på Forsterkede crnaer
for å undersøke size-bias-effekten på crnaer, ble første runde crnaer transkribert Av T7 RNA-polymerase og andre runde crnaer transkribert AV SP6 RNA-polymerase kjørt på 1,0% agarosegel (1 µg hver) og deretter blottet på nylonmembran (Biodyne® B Nylonmembran; PALL, East Hills, NY, USA). De ble hybridisert med 32P-merket SP6 opp primer (Tabell 1) og Ikke-(dT)18 (5′-GCGGCCGCTTTTTTTTTTTTTTTTTTTTTT-3′) oligonukleotid, henholdsvis. Ti picomoler av hver sonde ble merket MED ATP (omtrent 3000 Ci / mmol; Amersham Biosciences, Piscataway, NJ, USA) ved hjelp Av t4 polynukleotid kinase (Takara, Kyoto Japan). Etter hybridisering over natten i GMC-buffer (20) ved 45°C ble disse membranene grundig vasket i 1× standard saltvannscitrat (SSC)/1% SDS-oppløsning tre ganger ved romtemperatur. Analyse av hybridiserte signaler ble utført PÅ EN Bas 2000 Bildeanalysator (Fuji Photo Film, Tokyo, Japan)
Evaluering av cDNA-Biblioteker
Gelelektroforese. Fordeling av cDNA-innsatsstørrelser ble undersøkt ved elektroforese av crna syntetisert Av T7 RNA-polymerase ved Bruk Av noti-fordøyd MB-AL og MB-CL plasmid cdna som maler. Etter rensing ved hjelp Av Et Rneasy Mini-Sett ble crnaene elektroforesert på en formaldehydholdig agarosegel Med Perfekte RNA-Markører (EMD Biosciences, Madison, WI, USA). ETTER farging av gelen MED SYBR® Green II (Invitrogen) ble fluorescerende fargeintensitet analysert ved Hjelp Av ImageQuant® (versjon 5.0) Programvare På En FluorImager 595 (Amersham Biosciences).
Tilfeldig enkeltpass sekvenseringsanalyse. 3 ‘ – End sekvenser av cDNA kloner ble undersøkt VED DNA-sekvensering. Plasmid-DNA fra 768 tilfeldig plukket kloner ble renset ved hjelp AV EN Mfx-9600 Magnia® (Toyobo, Osaka, Japan) og analysert AV EN RISA 384-Kapillær Sequencer (Shimadzu, Kyoto, Japan) (16). Etter trimming av vektorsekvensen med Sequencher™ Programvareversjon 4.1 (Hitachi Software, Tokyo, Japan), ble de oppnådde cDNA-sekvensene som var lengre enn 200 nukleotidrester gruppert med cDNA-oppføringer I genbank® databasen og vår interne musedatabase. Undersøkelse av integriteten til 3 ‘endene av cDNA kloner ble utført ved en analyse av hvorvidt en kanonisk polyadenylering signal heksamer (5′-AATAAA-3’) eller dens enkelt-base varianter (11 signal heksamers) ble funnet innenfor 50 nukleotid rester oppstrøms fra poly(A) halen av cDNAs (21).
cDNA microarray. Microarray analyser ble utført for å sammenligne cDNA populasjoner I MB-AL og MB-CL biblioteker. Hjemmelagde cDNA nylon mikroarrays med 3534 prober ble utarbeidet, og radioisotopisk deteksjon ble utført (22). Kort sagt, cDNA plasmider, som ble isolert i vårt institutt og hvor hele sekvenser eller sluttsekvenser allerede var kjent, ble oppdaget Ved Hjelp Av GeneTAC™ RA1 (Genomic Solutions, Ann Arbor, MI, USA) På Biodyne b nylonmembran og deretter løst etter produsentens instruksjoner. Listen over cDNAs immobilisert på denne microarray er tilgjengelig på forespørsel fra forfatterne. MB-AL og MB-CL mål-cdnaer ble utarbeidet ved bruk av omvendt transkripsjon fra hver av de 1 µ cRNA-prøvene som ble brukt til innsettingsstørrelsesanalysen som beskrevet ovenfor. Disse målene ble fremstilt og merket i reaksjonsblandingen inneholder 1 µg av hver cRNA, 100 ng tilfeldig hexamer, 1× første-strand buffer, 10 mM DTT, hver 800 µM dATP, dTTP, og dGTP, 800 nM dCTP, 5 µL dCTP (>2500 Ci/mmol; Amersham Biovitenskap), og 100 U Hevet II RNase H – revers transkriptase ved romtemperatur i 10 minutter og deretter ved 42°C for 1 h. Etter alkaliske lyse og nøytralisering, merket cDNAs ble renset med QIAquick® PCR-rensing kit (QIAGEN) og telles. Målet cDNA ble utsatt for hybridisering med cDNA nylon microarray i nærvær av 1 µg polyadenylic syre og 2,5 µg Musen Sprinkelseng-1 DNA® (Invitrogen) i 250 µL PerfectHyb™ – buffer (Toyobo) på 68°C over natten. Etter streng vask ved 68°C med 2× SSC/1% SDS (to vasker på 15 min hver) og deretter med 0,1× SSC / 1% SDS (to vasker på 30 min hver), ble hybridiseringssignalene detektert og analysert på EN FLA-8000 (Fuji Fotofilm). CDNA-flekkene som viste sterkere signaler enn bakgrunnskontrollen ble valgt og utsatt for videre analyse. Etter å ha utført global normalisering, scatter plott av signalintensiteter av cDNA flekker som stammer FRA MB-AL og MB-CL mål ble oppnådd.
rna blot hybridisering. RNA blot hybridisering ble utført for å undersøke størrelse-bias effekter. MB-AL-og MB-CL-crnaene (hver 1,5 µ, syntetisert som beskrevet ovenfor) ble elektroforet på formaldehydholdig agarosegel og overført Til biodyne B nylonmembran. Sonden cDNAs ble fremstilt VED HJELP AV PCR-forsterkning. Primere ble utformet basert på sekvenser registrert I GenBank database eller vår in-house database; maler var cDNA plasmider som vi hadde isolert. Sonden av ribosomalt s6-protein var 724 bp i lengde, forsterket med 5 ‘- CGCTCGGCTGTGTCAAGATG-3 ‘og 5’ – GAGGACAGCCTCTCTGG-3 ‘primere, og sonden av varmesjokkprotein (hsp) 70, 940 bp i lengde, ble forsterket med 5′ – GATGGACAAGGCGCAGATCC-3′ og 5’-ctcgatggtgggtcctgagc – 3 ‘ primere. Disse sondene ble merket med dCTP (omtrent 3000 Ci / mmol; Amersham Biosciences) ved Hjelp Av RadPrime DNA Labeling System (Invitrogen). Etter over natten hybridisering I PerfectHyb buffer ved 65°C ble membranene vasket suksessivt med 0,1× SSC/1% SDS ved romtemperatur i 5 min og i 15 min, og deretter ved 65°C i 30 min. Hybridiseringssignalene ble detektert på EN bas 2000 bildeanalysator.
Resultater og diskusjon
cDNA-bibliotekets konstruksjonsmetode som vi utviklet i denne studien, er vist I Figur 1 (nylig introduserte trinn er uthevet med en stjerne). Denne metoden består av to runder med cRNA amplifikasjonstrinn, konvertering av crna til cdna, og rekombinasjonell kloning til et plasmid. Nøkkeltrinnet i denne metoden er adapter ligation å merke cDNA ender MED SP6 promoter sekvens. Når vi kan syntetisere cDNAs i dette formatet, blir det mulig å spesifikt reversere transkribere første runde cRNAs som inneholder SP6 promoter sekvensen på deres 3 ‘ ende. Den andre runde cRNAs syntetiseres MED SP6 RNA-polymerase ved hjelp av de resulterende dobbeltstrengede cDNAs som maler. Etter de to rundene AV rna-polymeraseassistert forsterkning, blir de resulterende crnaene deretter konvertert til dobbeltstrengede cdnaer ved revers transkripsjon ved hjelp av attB2-Not – (dT)18-primeren. Bruken av attB2-Not – (dT)18 primer i omvendt transkripsjon gjør det mulig for oss å konvertere bare crnaene som bærer sekvensen 5’-(A)18GCGGCCGC-3 ‘i deres 3’ ender. Fordi disse modifikasjonene bare skal tillate forsterkning og kloning av cdnaer som har riktig merkede ender, forventer vi at denne metoden vil minimere størrelsesforstyrrelser under rna-polymeraseassistert forsterkning.

trinnene i adapter-ligation-assistert cRNA forsterkning og følgende cDNA bibliotek konstruksjon er illustrert. For å opprettholde de forsterkede cDNA-størrelsene med de første runde cdnaene, introduserte vi noen modifikasjoner i konvensjonelle rna-polymeraseassisterte metoder (uthevet med stjerner). Blå og røde linjer indikerer henholdsvis cDNA og RNA. E. coli, Escherichia coli.
for å teste effekten av metoden konstruerte vi cDNA-biblioteker ved en konvensjonell metode (16) ved bruk av ikke-forsterket totalt rna fra musens hjerne (100 µ) og ved metoden beskrevet ovenfor ved bruk av 1 µ totalt RNA fra musens hjerne. Tabell 2 oppsummerer mengden cDNA og cRNA oppnådd i hvert trinn, beregnet som et gjennomsnitt av tre uavhengige eksperimentelle kjøringer AV MB-AL konstruksjon. Vi oppnådde i gjennomsnitt 6,4×107 uavhengige cDNA-kloner som den endelige utgangen. Siden dette antallet cDNA-kloner stammer fra totalt 36 ng cDNA generert ved hjelp av amplifikasjonsprotokollen, beregner vi at denne metoden gir ca. 1,2×1011 cDNA-kloner fra 1 µ totalt RNA.

for å undersøke størrelse-bias effekt under forsterkning trinn eksperimentelt, vi neste sammenlignet størrelsesfordelinger av første runde og andre runde cRNA VED RNA blot hybridisering analyse. I disse forsøkene ble første runde og andre runde cRNAs hybridisert med SP6 up primer og med Henholdsvis Ikke-(dT)18 oligonukleotid, fordi disse oligonukleotidprober oppdager bare cRNAs korrekt transkribert til slutten. Som vist i Figur 2 ble RNA-størrelsen på toppen av hybridiseringssignalet i andre runde cRNA funnet å være nesten det samme som i første runde cRNA, men med et lite skifte til mindre størrelse. Vi mistenker at avkorting av cRNAs trolig fant sted under andre runde cDNA syntese MED SP6 opp primer.

rna blot hybridisering ble utført for å undersøke størrelse-bias effekt på cRNAs. Hybridiseringssignaler langs retningen av cRNA-elektroforese ble plottet. RNA-størrelse ble bestemt basert på mobiliteten TIL rna-stigemarkøren. Blå linje, den første runde cRNA hybridisert MED SP6 opp probe; rød linje, den andre runde cRNA hybridisert Med Ikke-(dT)18 probe; PSL-verdi, signalintensitet angitt vilkårlig Av Bildemålingsprogramvaren (Fuji Photo Film).
for å vurdere kvaliteten på det resulterende forsterkede cDNA-biblioteket, sammenlignet vi også ulike funksjoner i cDNA-bibliotekene generert med og uten forsterkningen (HENHOLDSVIS MB-AL og MB-CL). Vi undersøkte først størrelsesfordelingen av cDNA-innsatsene, som vist I Figur 3A. resultatene av fluorescensintensitetssignaler oppnådd fra gelfargingsbildet indikerte at cDNA-innsatsstørrelsene MB-AL og MB-CL var like, men toppunktet var litt mindre I MB-AL (0.65 kb) sammenlignet med DET I MB-CL (0.8 kb). Deretter analyserte vi 3 ‘ – end-sekvenser av tilfeldig samplede cDNA-kloner fra hvert bibliotek. Clustering-resultatene for disse uttrykte sekvensmerkene (ESTs) er vist I Figur 3B og antyder at kompleksiteten TIL MB-AL var så høy SOM MB-CL. Det er imidlertid verdt å merke seg at svært redundante cDNA-kloner forsvant I MB-AL. Ved en statistisk analyse (Chi-test) ble det vist at forskjellen mellom redundanser MELLOM MB-AL og MB-CL var signifikant (sannsynligheten for at redundansene I MB-AL og MB-CL er like er <0,001). I tillegg undersøkte vi integriteten til 3 ‘ ender av cDNAs ved å analysere om HVER EST inneholdt et polyadenylasjonssignal heksamer eller ikke. Resultatene indikerte at 80,1% og 87,3% av cDNA-klonene I HENHOLDSVIS MB-al og MB-CL inneholdt plausible polyadenylasjonssignalsekvenser (21). Nedgangen i forekomsten av polyadenyleringssignalheksamerer I MB-AL indikerte sannsynligvis en økning i antall cDNA-kloner hvor cDNA-syntese ble internt primet på grunn av to runder med dt-priming.

ved mikroarray hybridisering undersøkte vi cDNA-populasjonen AV MB-AL i sammenligning MED MB-CL. Figur 3C viser et spredningsplott av hybridiseringssignaler for MB-AL og MB-CL-mål. Resultatene indikerte en betydelig høy korrelasjon av hybridiseringssignaler mellom MB-AL og MB-CL mål, noe som tyder på at det ikke var en skadelig effekt av forsterkningen i cDNA-populasjonen.
Til Slutt utførte VI rna blot hybridisering analyse for å undersøke størrelse-bias effekter med hensyn til housekeeping gener (Figur 3D). For ribosomalt s6-protein og hsp 70 var cRNA-størrelser både I MB-AL og MB-CL like og sammenlignbare med de som forventes fra deres rapporterte nukleotidsekvenser. Hybridiseringsmønsteret til hsp 70 cRNAs I MB-AL var imidlertid litt forskjellig fra DET I MB-CL, noe som indikerer at størrelsesbias-effekten ikke ble helt fjernet.metoden for cDNA bibliotek konstruksjon beskrevet i denne rapporten ble utviklet for å minimere størrelse skjevheter under forsterkning trinn assistert AV RNA polymerase. En SLIK RNA polymerase-assistert bibliotek byggemetode fra en liten RNA pool for omfattende sekvensering analyse har ikke blitt godt studert, mens cDNA / cRNA bibliotek byggemetoder for microarray hybridisering har vært aktivt forfulgt. Selv Om Lukyanov et al. (23) Og Piao et al. (24) rapporterte metoder som bruker PCR-forsterkning for å konstruere et cDNA-bibliotek fra en submikro-mengde av totalt RNA, HAR PCR-forsterkning i seg selv ulemper som alvorlig størrelse og populasjonsbias og lav gjengivelse av cDNA-forsterkning. Dette var grunnen til at vi prøvde å utvikle en cDNA bibliotek byggemetode basert PÅ RNA polymerase-assistert forsterkning i denne studien.
for dette formålet, utviklet vi en ny rna polymerase-assistert amplifikasjonsstrategi som vist I Figur 1 fordi Metodene For Eberwine et al. (5), Huang et al. (10), Og Lin et al. (11) har fortsatt ulemper for fremstilling av et plasmid-inneholdt cDNA egnet for omfattende genstrukturanalyse. I den tidligere er cDNA-syntese etter cRNA-forsterkning gjort med tilfeldige hexamers, og dette gjør de forsterkede cdnaene uunngåelig kortere enn de opprinnelige (5,7). I den sistnevnte, innføring av en homopolymere halen på den første-tråd cDNA slutten av terminal deoksynukleotidyl transferase (TdT) Eller Moloney murine leukemi virus (MMLV) revers transkriptase aktivitet (10,11) er fornuftig å minimere størrelse-bias effekt UNDER rna polymerase-assistert amplifikasjon som i vår metode. Imidlertid er homopolymeric tailing metoden kjent for å forårsake avkorting av cdna på grunn av uventet priming av cDNA syntese fra en homopolymeric strekning av interne RNA-sekvenser. Fordi adapterligasjonsmetoden beskrevet i denne studien kunne gi en bestemt sekvens for cDNA-priming, kan vi forvente å redusere omfanget av cDNA-trunkering ved hjelp av vår metode.i prinsippet bør vår metode bare forsterke cdna flankert av en iboende poly (A) hale og SP6 adapter sekvens, som ble generert i første runde cDNA syntese. Resultatene vist I Figur 2 OG FIGUR 3A var i hovedsak i samsvar med dette, selv om cDNA-populasjonen i det mindre størrelsesområdet var litt økt. Resultatene AV rna blot-analyse (Figur 3D) viste også at størrelsesbias-effekten kunne reduseres, men ikke helt fjernet selv i vår metode. Dette var sannsynligvis fordi avkortede cdnaer lagt til av en poly (A) hale og SP6 promoter-sekvensen ble generert under konvertering av cRNA til cDNA. Dette synet ble støttet av det faktum at forekomsten rate av polyadenylering signal sekvenser I MB-AL var signifikant lavere enn det som er observert i den konvensjonelle cDNA bibliotek (21). Avkorting av cdna kan oppstå under cDNA-syntese på grunn av intern priming AV RNA med dt-tailed primer og SP6 primer. Fordi homopolymere strekninger forekommer ofte i eukaryote mrna, kan homopolymere tailing metoden pålegge mer alvorlig størrelse skjevhet på flere runde cRNA forsterkning enn vår adapter-ligation metoden. Selv om økning av reaksjonstemperaturen under revers transkripsjon og reduksjon av primerkonsentrasjonen kan undertrykke den avvikende interne priming i cDNA-syntese til en viss grad, er det kjent å være vanskelig å eliminere dette helt i dag. Således, selv om vi utførte to-runde cRNA-forsterkning for demonstrasjon av den generelle protokollen, anbefaler vi i praksis å hoppe over den andre runde cRNA-forsterkningen når nok cRNA kan oppnås i første runde-reaksjonen.Med Tanke på disse resultatene konkluderer vi med at vår nye metode er nyttig for cDNA-bibliotekskonstruksjon fra en liten mengde start-RNA. Faktisk har vi vellykket og rutinemessig brukt denne metoden for bygging av cDNA-biblioteker når mengden av totalt RNA er mindre enn 1 µ (data ikke vist). Fordi 1 µ totalt RNA kunne gjenvinnes fra 105-106 pattedyrceller eller 1 mg pattedyrvev, kan cDNA-biblioteker enkelt konstrueres fra celler fraksjonert av cellesorterere eller mikrodisseksjon ved hjelp av vår metode. Videre, fordi vi beregner at denne metoden gjør det mulig for oss å oppnå mer enn 105 cDNA-kloner fra 1 pg totalt RNA, som er nær mengden av totalt RNA i en enkelt celle, kan et enkelt celle-avledet cDNA-bibliotek bli konstruert basert på denne adapterligasjon-assistert cRNA-forsterkning.
Takk
forfatterne er takknemlige for instruksjon av statistisk analyse Fra Dr. Kazuharu Misawa; divisjon av plasmid DNAs Fra Dr. Hisashi Koga; og teknisk hjelp Fra Ms. Akiko Ukigai-Ando Og Mr. Takashi Watanabe. Studien ble støttet av et stipend fra Kazusa DNA Research Institute Til R. O., R. F. K. og O. o.
Konkurrerende Interesser Statement
forfatterne erklærer ingen konkurrerende interesser.
- 1. I. k. k., a. K. K., a. K. K., a. K. K. K., et al.. 1996. Høy effektivitet full-lengde cDNA kloning av biotinylert CAP trapper. Genomikk 37:327-336.Cas, Google Scholar
- 2. I tillegg til å være en del av det meste av verden, er det en stor del av verden. 1997. Konstruksjon og karakterisering av menneskelige hjerne cDNA biblioteker egnet for analyse av cDNA kloner koding relativt store proteiner. DNA-Res. 4:53-59.Crossref, Medline, CAS, Google Scholar
- 3. I tillegg til å være en av de mest kjente i verden. 1997. Konstruksjon og karakterisering av en full lengde-beriket og en 5′-end-beriket cDNA bibliotek. Gen 200: 1149-1156.Iversen, Google Scholar
- 4. Ohara, O. Og G. Temple. 2001. Retningsbestemt cDNA bibliotek konstruksjon assistert av in vitro rekombinasjon reaksjon. Nukleinsyrer Res. 29: e22.Crossref, Medline, CAS, Google Scholar
- 5. Det er en av de mest kjente artene i verden. 1992. Analyse av genuttrykk i enkelt levende nevroner. Proc. Natl. Acad. Sci. USA 89: 3010-3014.Crossref, Medline, CAS, Google Scholar
- 6. Han er en av de eldste i verden. 2000. High-fidelity mRNA-forsterkning for genprofilering. Nat. Bioteknologi. 18:457–459.Crossref, Medline, CAS, Google Scholar
- 7. Baugh, L. R., A. A. Hill, E. L. Brown og C. P. Hunter. 2001. Kvantitativ analyse av mRNA amplifikasjon ved in vitro transkripsjon. Nukleinsyrer Res. 29: e29.Crossref, Medline, CAS, Google Scholar
- 8. A. V. chenchik, Og P. D. Siebert. 2001. BRUK AV SMART™ – generert cDNA for genuttrykksstudier i flere humane svulster. Bioteknologi 30:158-163.Link, CAS, Google Scholar
- 9. G., r., R. Silva, B. S. Stolf, E. B. Cristo, R. Hirata, Jr., F. A. Soares, L. Reis, E. J. Neves, et al.. 2003. Komparativ analyse av forsterket OG ikke-forsterket RNA for hybridisering i cDNA microarray. Anal. Biochem. 321:244–251.Crossref, Medline, CAS, Google Scholar
- 10. Han er en av de eldste i verden. 2003. Avkortet Bcl-2, en potensiell pre-metastisk markør i prostatakreft. Biochem. Biophys. Res. Commun. 306:912–917.Crossref, Medline, CAS, Google Scholar
- 11. Lin, S. L. Og S. Y. Ying. 2003. mRNA / cDNA bibliotek konstruksjon ved HJELP AV RNA-polymerase sykling reaksjon. Metoder Mol. Biol. 221:129–143.Cas, google Scholar
- 12. Matz, M. V. 2003. Forsterkning av representative cDNA-bassenger fra mikroskopiske mengder animalsk vev. Metoder Mol. Biol. 221:103–116.Cas, google Scholar
- 13. I tillegg til å være en del av det norske samfunnet, er det en del av det norske samfunnet. 2003. Global forsterkning av mRNA ved mal-bytte PCR: linearitet og søknad til microarray analyse. Nukleinsyrer Res. 22: e142.Scholar
- 14. Polacek, D. C., A. G. Passerini, C. Shi, N. M. Francesco, E. Manduchi, G. R. Grant, S. Powell, H. Bischof, et al.. 2003. Fidelity og forbedret følsomhet for differensial transkripsjon profiler etter lineær forsterkning av nanogram mengder endotel mRNA. Physiol. Genomikk 13:147-156.Crossref, Medline, CAS, Google Scholar
- 15. Wilson, C., S. D. Pepper, Y. Hey Og C. J. Miller. 2004. Amplifikasjonsprotokoller introduserer systematiske, men reproduserbare feil i genuttrykksstudier. Bioteknologi 36:498-506.Link, CAS, Google Scholar
- 16. O., T. Nagase, G. A. k., a. k. K., a. K. K., a. K., a. K., a. K., a. K., a. K., a. k., a. k., a. k., a. k., a. k., et al.. 2002. Karakterisering av størrelse-fraksjonert cDNA bibliotek generert av in vitro rekombinasjon-assistert metode. DNA Res. 9:47-57.Crossref, Medline, CAS, Google Scholar
- 17. Ohara, O. 2003. Konstruksjon av størrelsesfraksjonert cDNA-bibliotek assistert av en in vitro rekombinasjonsreaksjon. Metoder Mol. Biol. 221:59–71.Cas, google Scholar
- 18. Gubler, U. Og B. J. Hoffman. 1983. En enkel og svært effektiv metode for å generere cDNA-biblioteker. Gen 25: 263-269.Crossref, Medline, CAS, Google Scholar
- 19. Sambrook, J. Og D. W. Russell. 2001. Fremstilling av plasmid DNA ved alkalisk lysis MED SDS, s. 1.31–1.42. Molekylær Kloning, (3. utgave). Hotell I Nærheten Av Csh Laboratory Press, Cold Spring Harbor, NYGoogle Scholar
- 20. Church, G. M. Og W. Gilbert. 1984. Genomisk sekvensering. Proc. Natl. Acad. Sci. USA 81: 1991-1995.Crossref, Medline, CAS, Google Scholar
- 21. Det er en av de mest kjente artene i verden. 2000. Mønstre av variant polyadenylering signalbruk i humane gener. Genom Res. 10:1001-1010.Crossref, Medline, CAS, Google Scholar
- 22. T. s., s. A. Jaradat, M. K. Lim, G. J. Kargul, X. Wang, M. J. Grahovac, S. Pantano, Y. Sano, et al.. 2000. Genom-wide expression profilering av mid-svangerskap placenta og embryo ved hjelp av en 15.000 mus utviklings cDNA microarray. Proc. Natl. Acad. Sci. USA 97: 9127-9132.Crossref, Medline, Google Scholar
- 23. I tillegg til dette, er det et stort antall av dem. 1997. Konstruksjon av cDNA-biblioteker fra sma mengder total RNA ved hjelp AV supresjon PCR-effekten. Biochem. Biophys. Res. Commun. 230:285–288.Crossref, Medline, CAS, Google Scholar
- 24. Piao, Y., N. T. Ko, M. K. Lim, Og M. Ko. 2001. Bygging av lange transkripsjoner beriket cDNA biblioteker fra submicrogram mengder av totale Rna av en universell PCR forsterkning metode. Genom Res. 11:1553-1558.Google Scholar
