なぜ遺伝子発現のCap解析を開発するのですか?
Cap trapper法、トレハロースの使用、正規化/減算法、新しいクローニングベクターから始まった1990年代後半のPiero Carninciと林崎義秀による研究は、遺伝子発現のCap解析の開発の段階を設定した。 キャップトラッパーでは、全長cDNA/mRNAハイブリッドが単離され、mRNAはキャップ構造上に化学的にビオチン化され、ストレプトアビジン被覆磁気ビーズは雑種を捕獲する。 この一連の主要なDNA技術の進歩は、自然界ではマイルストーン5として記述されています(www.nature.com/milestones/miledna/full/miledna05.html CAGEの開発における彼らの目的は、ヒト転写開始部位とそのプロモーターの大部分を包括的にマッピングするための技術を作成することでした。 実際、各プロモーター部位での遺伝子転写の活性をプロファイルする技術は、CAGEが現場に登場するまで存在しませんでした。 メッセンジャー RNA(mRNA)は、ゲノム上の個々の遺伝子にコードされた情報と、生物の運命を決定するタンパク質の構成との間の重要なリンクを表します。 私たちは自分自身に尋ねました:遺伝子とそのユニークな異なるRnaの特定の発現を駆動するゲノム領域、またはプロモーターは何ですか? 実際、完全長cDNA収集は、ほとんどの遺伝子が複数の転写開始部位を有することを示しており、したがって、転写産物の様々な形態の発現を担う制御領域、すなわちプロモーターを同定することは非常に困難である。
“転写”と呼ばれるこの情報伝達プロセスの複雑さを分析するには、遺伝子発現の定性的側面と定量的側面の両方を捉えることができる洗練された分子ツールの開発が必要である。 そのため、独自のゲノムワイド転写開始検出技術であるCAGEでは、プロモーター使用解析を含む組織/細胞/条件特異的転写開始部位(TSS)の同時同定により、遺伝子発現プロファイリングを行うことができます。 CAGEは、分析されたサンプル中のmRNAの元の濃度(RNA頻度)を反映する5’末端mrnaからの最初の20ヌクレオチドから導出されるDNAタグのコンカタマーの調製およ
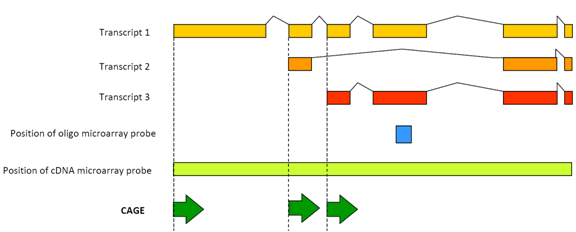
図1:CAGEは、各プロモーター転写産物の転写活性を検出します。
発現配列タグ(ESTs)は、ヒトゲノム上にそれらを整列させることによってプロモーター要素を同定するための技術開発の初期段階で使用されました。 しかしながら、このプロセスは、物理的なcDNAおよびSanger配列決定を処理するコストのために非常に高価である。 これらの問題を克服するための一つの方法は、ESTシーケンシングよりも一桁以上の感度で転写物を検出し、徹底的に転写物を同定し、そのプロモーターを同定し、遺伝子発現のデジタル尺度としてタグをカウントすることにより、発現プロファイルとそれらを相関させるために開発されたタグ付け技術を使用することである。
これらの配列(”タグ”とも呼ばれる)は、単純な計算手順(BLASTと呼ばれる)によってゲノム配列に整列され、カウントされ、RNA発現の頻度の測定が得られる。 これらの配列タグは、RNA転写の開始部位を同定するので、それらはまた、それらの開始部位に近いゲノム配列を同定する。 隣接する領域は、マウスからヒトまで、多くの複雑な生物で遭遇する多くの異なる条件で遺伝子を転写させるゲノム配列であるコアプロモータ領域で
ケージの属性
ケージは、古典的なマイクロアレイベースの発現検出技術に比べて大きな利点を持っています。 実際には、それぞれの生物現象、組織、細胞などにおいてRNA転写を引き起こすプロモーターを同定することによって、 我々は、分析されたサンプルで発現されているRNAアイソフォームのプロモーターにある配列を見ることによって、各生物現象に特異的なDNA調節要素を同定す プロモーターは、転写因子(TF)と呼ばれるそれらの結合タンパク質によって認識され、転写を促進または抑制する特定の配列、または転写因子結合部位(TFBS) 計算方法を用いて、我々の研究者は、TFBのための同様の発現プロファイルを有するプロモーターを分析し、ゲノムの転写出力を担当するTFsを同定する。 遺伝子内の各プロモーターのCAGEタグの数を数えることにより、RNA発現レベル(これは頻度のデジタル検出である)だけでなく、重要なことに、RNAが転写される様々な代替プロモーターのうちのどれからかを決定することができるようになった。
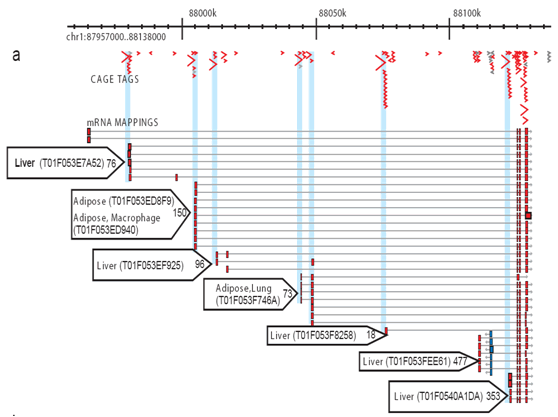
図2:CAGEは、各プロモーターサイトでの活性化の包括的なプロファイリングを可能にします。 各ライブラリーについて、多数のCAGEタグが配列決定され、ゲノムに整列され、各プロモーターにおける特異的転写活性を測定し、各プロモーターの寄与を区別することができる。 この単純化された例は、脂肪および肝臓コアプロモーターを示す。 小さい青い矢:個々のおりの札;赤い矢:ティッシュのための促進者の使用法の好み;赤い箱:中心の促進者の地域。言及されたように、CAGEは、cDNAの5’末端を捕捉するための最初のステップとしてcap−trapingを使用し、次いで、mRNA Tssに対応する2 0〜2 7ntの短い配列(tag)に形質転換される。 私達は私達が第二世代の配列を使用するdeepCAGEに最近移るまでSangerの配列との連結されたおりの札を使用して何百万のマウスおよび人間のおりの札を、作
2006年までは、90年代後半に建設されたオリジナルのrisaシーケンシング-パイプラインでケージ-ライブラリーを運営していましたが、島津製作所と共同で開発した384個のキャピラリーの配列を持つ唯一のキャピラリー-シーケンサーを含んでいました。
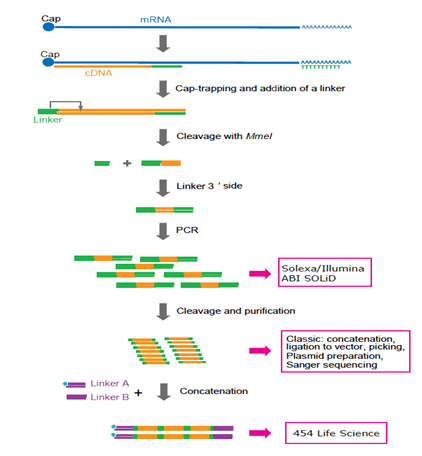
図3:様々なプラットフォームに適応ケージ準備プロトコルの表現。 今SolexaおよびIlluminaは好まれます。 454Life Sciences(FLX system)は、連結に追加のPCRサイクルと複雑な操作が必要なため、もはや使用されていません。 将来的には、PCRが必要とされない可能性があるため、単一分子配列決定技術が好まれるであろう。
タグ付けとシーケンス技術は、遺伝子解析のシリアル分析(SAGE)法を使用して開発されていますが、CAGEは、キャップサイト-オールmrnaと非コードRnaの大部分を運 私達は地図を描く効率を高めるために27nt長いおりの札を開発しました。
