Obiettivi di Apprendimento
- Descrivere la struttura fondamentale di un amminoacido
- Descrivere la struttura chimica delle proteine
- Riassumere le caratteristiche uniche di proteine
All’inizio di questo capitolo, un famoso esperimento è stato descritto, in cui scienziati sintetizzato aminoacidi in condizioni che simulano quelle presenti sulla terra molto prima che l’evoluzione della vita come noi la conosciamo. Questi composti sono in grado di legarsi tra loro essenzialmente in qualsiasi numero, producendo molecole di qualsiasi dimensione che possiedono una vasta gamma di proprietà fisiche e chimiche e svolgono numerose funzioni vitali per tutti gli organismi. Le molecole derivate dagli amminoacidi possono funzionare come componenti strutturali delle cellule e delle entità subcellulari, come fonti di nutrienti, come serbatoi di accumulo di atomi ed energia e come specie funzionali come ormoni, enzimi, recettori e molecole di trasporto.
Amminoacidi e legami peptidici

Figura 1.
Un amminoacido è una molecola organica in cui un atomo di idrogeno, un gruppo carbossilico (–COOH) e un gruppo amminico (–NH2) sono tutti legati allo stesso atomo di carbonio, il cosiddetto carbonio α. Il quarto gruppo legato al carbonio α varia tra i diversi amminoacidi ed è chiamato residuo o catena laterale, rappresentato nelle formule strutturali dalla lettera R. Un residuo è un monomero che risulta quando due o più amminoacidi si combinano e rimuovono le molecole d’acqua. La struttura primaria di una proteina, una catena peptidica, è costituita da residui di aminoacidi. Le caratteristiche uniche dei gruppi funzionali e dei gruppi R consentono a questi componenti degli amminoacidi di formare legami idrogeno, ionici e disolfuro, insieme alle interazioni polari/non polari necessarie per formare strutture proteiche secondarie, terziarie e quaternarie. Questi gruppi sono composti principalmente da carbonio, idrogeno, ossigeno, azoto e zolfo, sotto forma di idrocarburi, acidi, ammidi, alcoli e ammine. Alcuni esempi che illustrano queste possibilità sono forniti nella Figura 1.
Gli amminoacidi possono legarsi chimicamente insieme per reazione del gruppo acido carbossilico di una molecola con il gruppo amminico di un altro. Questa reazione forma un legame peptidico e una molecola d’acqua ed è un altro esempio di sintesi di disidratazione (Figura 2). Molecole formate collegando chimicamente numeri relativamente modesti di aminoacidi (circa 50 o meno) sono chiamati peptidi, e prefissi sono spesso utilizzati per specificare questi numeri: dipeptidi (due aminoacidi), tripeptidi (tre aminoacidi), e così via. Più in generale, il numero approssimativo di aminoacidi è designato: gli oligopeptidi si formano unendo fino a circa 20 aminoacidi, mentre i polipeptidi sono sintetizzati fino a circa 50 aminoacidi. Quando il numero di amminoacidi collegati tra loro diventa molto grande, o quando più polipeptidi vengono utilizzati come subunità di costruzione, le macromolecole che risultano sono chiamate proteine. La lunghezza continuamente variabile (il numero di monomeri) di questi biopolimeri, insieme alla varietà di possibili gruppi R su ciascun amminoacido, consente una diversità quasi illimitata nei tipi di proteine che possono essere formate.

Figura 2. La formazione del legame peptidico è una reazione di sintesi di disidratazione. Il gruppo carbossilico del primo amminoacido (alanina) è collegato al gruppo amminico del secondo amminoacido in entrata (alanina). Nel processo, viene rilasciata una molecola d’acqua.
Pensaci
- Quanti aminoacidi ci sono nei polipeptidi?
Struttura proteica
La dimensione (lunghezza) e la sequenza aminoacidica specifica di una proteina sono i principali determinanti della sua forma e la forma di una proteina è fondamentale per la sua funzione. Ad esempio, nel processo di fissazione biologica dell’azoto (vedi Cicli biogeochimici), i microrganismi del suolo noti collettivamente come rhizobia interagiscono simbioticamente con le radici di piante leguminose come soia, arachidi o fagioli per formare una nuova struttura chiamata nodulo sulle radici delle piante. La pianta produce quindi una proteina portante chiamata leghemoglobina, una proteina che trasporta azoto o ossigeno. Leghemoglobin lega con un’affinità molto alta al suo ossigeno del substrato ad una regione specifica della proteina in cui la forma e la sequenza dell’amminoacido sono appropriate (il sito attivo). Se la forma o l’ambiente chimico del sito attivo è alterata, anche leggermente, il substrato potrebbe non essere in grado di legarsi con la stessa forza o potrebbe non legarsi affatto. Pertanto, affinché la proteina sia pienamente attiva, deve avere la forma appropriata per la sua funzione.
La struttura proteica è classificata in termini di quattro livelli: primario, secondario, terziario e quaternario. La struttura primaria è semplicemente la sequenza di aminoacidi che compongono la catena polipeptidica. La figura 3 descrive la struttura primaria di una proteina.
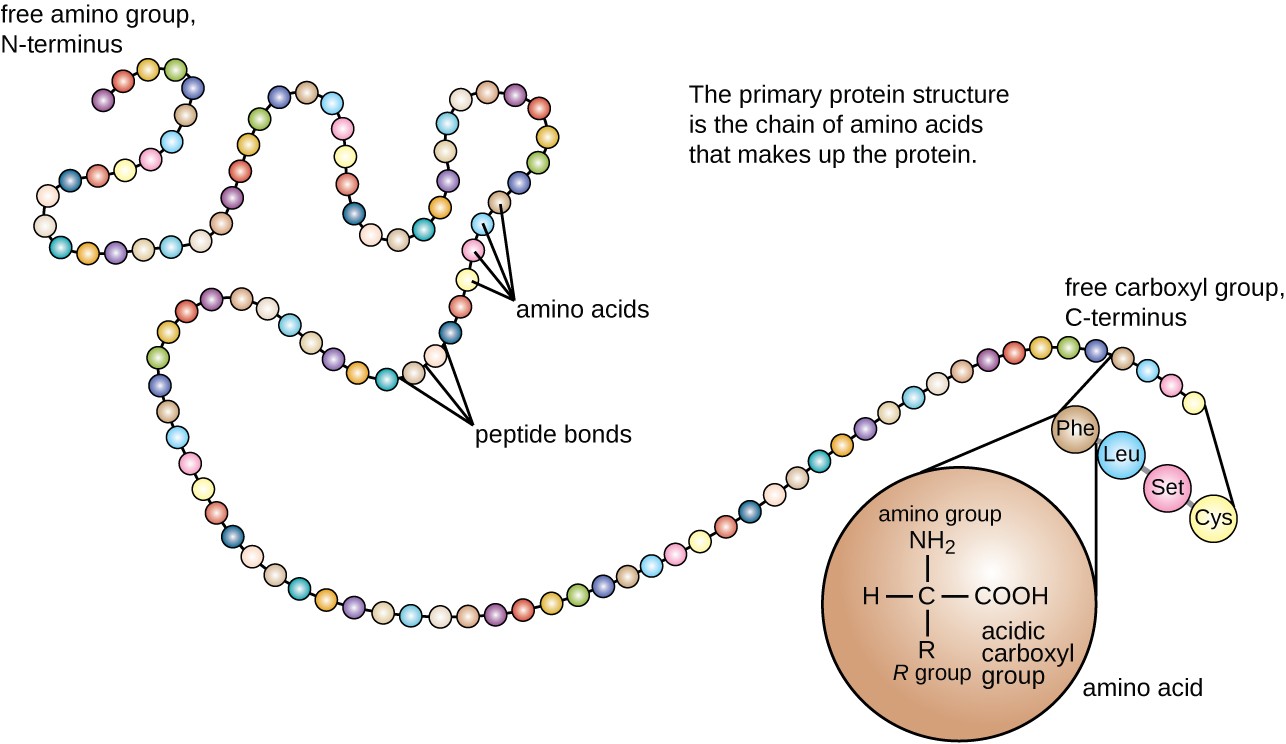
Figura 3. Fare clic per visualizzare un’immagine ingrandita. La struttura primaria di una proteina è la sequenza di aminoacidi. (credit: modifying of work by National Human Genome Research Institute)
La catena di aminoacidi che definisce la struttura primaria di una proteina non è rigida, ma invece è flessibile a causa della natura dei legami che tengono insieme gli amminoacidi. Quando la catena è sufficientemente lunga, il legame dell’idrogeno può accadere fra l’ammina ed i gruppi funzionali carbonilici all’interno della spina dorsale del peptide (escluso il gruppo laterale di R), con conseguente piegatura localizzata della catena del polipeptide nelle eliche e nei fogli. Queste forme costituiscono la struttura secondaria di una proteina. Le strutture secondarie più comuni sono il foglio α-elica e β-pieghettato. Nella struttura α-elica, l’elica è tenuta da legami idrogeno tra l’atomo di ossigeno in un gruppo carbonilico di un amminoacido e l’atomo di idrogeno del gruppo amminico che è solo quattro unità di amminoacidi più lontano lungo la catena. Nel foglio β-pieghettato, le pieghe sono formate da legami idrogeno simili tra sequenze continue di gruppi carbonilici e amminici che sono ulteriormente separati sulla spina dorsale della catena polipeptidica (Figura 4).

Figura 4. La struttura secondaria di una proteina può essere una α-elica o un foglio β-pieghettato, o entrambi.
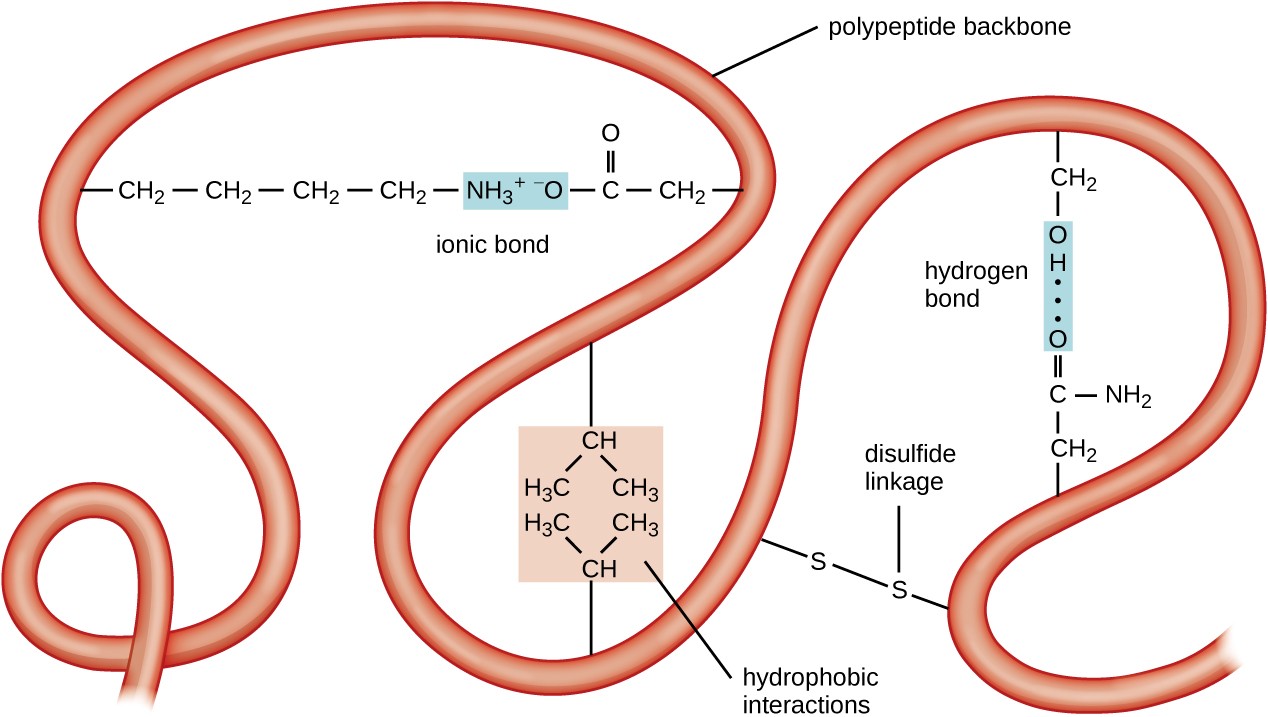
Figura 5. Clicca per ingrandire l’immagine. La struttura terziaria delle proteine è determinata da una varietà di forze attrattive, tra cui interazioni idrofobiche, legame ionico, legame idrogeno e legami disolfuro.
Il livello successivo di organizzazione proteica è la struttura terziaria, che è la forma tridimensionale su larga scala di una singola catena polipeptidica. La struttura terziaria è determinata dalle interazioni fra i residui dell’amminoacido che sono distanti nella catena. Una varietà di interazioni danno origine alla struttura terziaria della proteina, come i ponti disolfuro, che sono legami tra i gruppi funzionali sulfidrilici (–SH) sui gruppi laterali degli amminoacidi; legami idrogeno; legami ionici; e interazioni idrofobiche tra catene laterali non polari. Tutte queste interazioni, deboli e forti, si combinano per determinare la forma tridimensionale finale della proteina e la sua funzione (Figura 5).
Il processo mediante il quale una catena polipeptidica assume una forma tridimensionale su larga scala è chiamato ripiegamento proteico. Si dice che le proteine piegate che sono pienamente funzionali nel loro normale ruolo biologico posseggano una struttura nativa. Quando una proteina perde la sua forma tridimensionale, potrebbe non essere più funzionale. Queste proteine spiegate sono denaturate. La denaturazione implica la perdita della struttura secondaria e della struttura terziaria (e, se presente, della struttura quaternaria) senza la perdita della struttura primaria.
Alcune proteine sono assemblaggi di diversi polipeptidi separati, noti anche come subunità proteiche. Queste proteine funzionano adeguatamente solo quando tutte le subunità sono presenti e opportunamente configurate. Le interazioni che tengono insieme queste subunità costituiscono la struttura quaternaria della proteina. La struttura quaternaria complessiva è stabilizzata da interazioni relativamente deboli. L’emoglobina, ad esempio, ha una struttura quaternaria di quattro subunità proteiche globulari: due polipeptidi α e due β, ciascuno contenente un eme a base di ferro (Figura 6).

Figura 6. Una molecola di emoglobina ha due polipeptidi α e due β insieme a quattro gruppi eme.
Un’altra importante classe di proteine sono le proteine coniugate che hanno una porzione non proteica. Se la proteina coniugata ha un carboidrato attaccato, è chiamata glicoproteina. Se ha un lipide attaccato, si chiama lipoproteina. Queste proteine sono componenti importanti delle membrane. La figura 7 riassume i quattro livelli di struttura proteica.

Figura 7. La struttura proteica ha quattro livelli di organizzazione. (credito: modifica del lavoro del National Human Genome Research Institute)
Pensaci
- Cosa può accadere se la struttura primaria, secondaria, terziaria o quaternaria di una proteina viene modificata?
Struttura primaria, proteine disfunzionali e fibrosi Cistica

Figura 8. Fare clic per visualizzare un’immagine ingrandita. La normale proteina CFTR è una proteina canale che aiuta il sale (cloruro di sodio) muoversi dentro e fuori delle cellule.
Le proteine associate alle membrane biologiche sono classificate come estrinseche o intrinseche. Le proteine estrinseche, chiamate anche proteine periferiche, sono vagamente associate a un lato della membrana. Le proteine intrinseche, o proteine integrali, sono incorporate nella membrana e spesso funzionano come parte dei sistemi di trasporto come proteine transmembrana. La fibrosi cistica (CF) è una malattia genetica umana causata da un cambiamento nella proteina transmembrana. Colpisce soprattutto i polmoni, ma può anche influenzare il pancreas, fegato, reni e intestino. La CF è causata da una perdita dell’amminoacido fenilalanina in una proteina transmembrana della fibrosi cistica (CFTR). La perdita di un amminoacido modifica la struttura primaria di una proteina che normalmente aiuta a trasportare sale e acqua dentro e fuori le cellule (Figura 8).
Il cambiamento nella struttura primaria impedisce alla proteina di funzionare correttamente, il che fa sì che il corpo produca muco insolitamente denso che intasa i polmoni e porta all’accumulo di muco appiccicoso. Il muco ostruisce il pancreas e impedisce agli enzimi naturali di aiutare il corpo ad abbattere il cibo e assorbire i nutrienti vitali.
Nei polmoni di individui con fibrosi cistica, il muco alterato fornisce un ambiente in cui i batteri possono prosperare. Questa colonizzazione porta alla formazione di biofilm nelle piccole vie aeree dei polmoni. I patogeni più comuni trovati nei polmoni dei pazienti con fibrosi cistica sono Pseudomonas aeruginosa (Figura 9a) e Burkholderia cepacia. Pseudomonas si differenzia all’interno del biofilm nel polmone e forma grandi colonie, chiamate Pseudomonas “mucoidi”. Le colonie hanno una pigmentazione unica che si presenta nei test di laboratorio (Figura 9b) e fornisce ai medici il primo indizio che il paziente ha CF (tali colonie sono rare in individui sani).

Figura 9. (a) Un micrografo elettronico a scansione mostra il batterio opportunistico Pseudomonas aeruginosa. (b) Pigmento che produce P. aeruginosa su cetrimide agar mostra il pigmento verde chiamato piocianina. (credito a: modifica del lavoro da parte dei Centers for Disease Control and Prevention)
Concetti chiave e sommario
- Gli aminoacidi sono piccole molecole essenziali per tutta la vita. Ognuno ha un carbonio α a cui sono legati un atomo di idrogeno, un gruppo carbossilico e un gruppo amminico. Il quarto gruppo legato, rappresentato da R, varia in composizione chimica, dimensione, polarità e carica tra diversi amminoacidi, fornendo variazioni nelle proprietà.
- I peptidi sono polimeri formati dal legame degli amminoacidi tramite la sintesi di disidratazione. I legami tra gli amminoacidi collegati sono chiamati legami peptidici. Il numero di aminoacidi collegati tra loro può variare da pochi a molti.
- Le proteine sono polimeri formati dal legame di un numero molto elevato di amminoacidi. Svolgono molte importanti funzioni in una cellula, fungendo da nutrienti ed enzimi; molecole di stoccaggio per carbonio, azoto ed energia; e componenti strutturali.
- La struttura di una proteina è un determinante critico della sua funzione ed è descritta da una classificazione graduata: primaria, secondaria, terziaria e quaternaria. La struttura nativa di una proteina può essere interrotta dalla denaturazione, con conseguente perdita della sua struttura di ordine superiore e della sua funzione biologica.
- Alcune proteine sono formate da diverse subunità proteiche separate, l’interazione di queste subunità che compongono la struttura quaternaria del complesso proteico.
- Le proteine coniugate hanno una porzione non polipeptidica che può essere un carboidrato (formando una glicoproteina) o una frazione lipidica (formando una lipoproteina). Queste proteine sono componenti importanti delle membrane.
Scelta multipla
Quale dei seguenti gruppi varia tra i diversi amminoacidi?
- atomo di idrogeno
- gruppo carbossilico
- gruppo R
- gruppo amminico
Gli amminoacidi presenti nelle proteine differiscono in quale dei seguenti?
- dimensioni
- forma
- gruppi laterali
- tutti i sopra
Quali dei seguenti legami non sono coinvolti nella struttura terziaria?
- i legami peptidici
- legami ionici
- interazioni idrofobiche
- legami a idrogeno
Riempire il vuoto
La sequenza di aminoacidi in una proteina è chiamata its __________.
La denaturazione implica la perdita del __________ e __________ strutture senza la perdita dell’__________ struttura.
True/False
Un cambiamento in un amminoacido in una sequenza proteica provoca sempre una perdita di funzione.
Pensaci
- Riscaldare sufficientemente una proteina può causarne la denatura. Considerando la definizione di denaturazione, cosa dice questa affermazione sui punti di forza dei legami peptidici rispetto ai legami idrogeno?
- L’immagine mostrata rappresenta un tetrapeptide.

- Quanti legami peptidici sono in questa molecola?
- Identificare i gruppi laterali dei quattro aminoacidi che compongono questo peptide.
