Úvod
kompletní sekvence genomu různých organismů, včetně savců, se nedávno stal k dispozici jako následek rychlý pokrok v sekvenování DNA technologie. Zejména u savců však analýza transkriptů stále hraje klíčovou roli při překlenutí mezery mezi genomem a proteomem. Je to hlavně proto, že v současné době, nemůžeme přesně předpovědět struktury přepisy odvozený od konkrétního genu z genomické informace sám. Tím pádem, jako metoda pro analýzu transkriptů, konstrukce knihovny cDNA je zásadní, dokonce i v éře sekvenování po genomu. I když klonování cDNA genů zájmu pomocí PCR poskytl zjednodušený alternativní trasy analyzovat přepis struktury bez cDNA knihovna výstavby, konstrukce cDNA knihovny je přístup na výběr, když se velký počet cDNAs z jediné mRNA zdroje mají být analyzovány. K dnešnímu dni bylo vynaloženo velké úsilí na vývoj metody přípravy vysoce kvalitních knihoven cDNA (1-4). Naproti tomu otázka, jak připravit vysoce kvalitní knihovnu cDNA z malého bazénu RNA, nebyla tak aktivně řešena. Nicméně, to se stalo vysoce prioritní cíl, protože vědci jsou často zájem o hypotetické geny, které jsou předpověděl být vyjádřena pouze v některých typech buněk nebo tkání za zvláštních podmínek, jako jsou ty, které viděl v patologických vzorků.
existuje řada zpráv popisujících použití malého množství zdrojové RNA pro generování amplifikované cDNA nebo cRNA, z nichž lze připravit cíle pro analýzu microarray (5-15). Jejich konečným cílem je získat plnou délku, neobjektivní RNA, která je vysoce reprezentativní pro původní mRNA. K provedení tohoto, tyto metody obvykle přijmout PCR nebo in vitro transkripce pomocí T7 RNA polymerázy pro amplifikaci původní mRNA v podobě cDNA nebo cRNA, resp. I když srovnání projevu profilů je jedním z nejvíce efektivní přístupy zkoumat rozdíly ve fyziologických stavů buněk nebo tkání, strukturní charakterizaci přepisy (např. identifikace sestřihových vzory, transkripce začátek stránky a přepis ukončení stránky) nemůže být provedeno tak microarray analýzy. Sekvenční analýza každého přepisu je v těchto případech nevyhnutelná.
vyvinuli jsme metodu, která umožňuje použít malé množství počáteční RNA k vytvoření knihovny cDNA vhodné pro klonování genů a komplexní sekvenační analýzu. Metoda značí 3′ a 5 ‚ konce cDNA prvního kola sekvencemi promotoru fágu T7 a SP6, aby se minimalizovaly účinky zkreslení velikosti během amplifikace. Po dvoukolové amplifikaci cRNA jsme přeměnili amplifikované Crna na cDNA, klonovali tyto produkty na plazmid a poté vyhodnotili výslednou knihovnu cDNA ve srovnání s knihovnou vytvořenou konvenční metodou (4,16,17).
materiály a metody
Crna amplifikace
sekvence oligonukleotidů použité v této studii jsou uvedeny v tabulce 1. Pro stanovení protokolu jsme použili 1 µg celkové RNA připravené z mozku myši ICR (8týdenní samci myší) jako šablonu. Směs(10 µL) 1 µg celkové RNA a 100 pmol T7-Not-(dT)18 primeru byla inkubována při 70°C po dobu 10 minut a poté se ochladila na ledu. Dvouvláknová syntéza cDNA a ligace adaptérů byly provedeny podle protokolů SUPERSCRIPT® Plasmid System (Invitrogen, Carlsbad, CA, USA) (4,16,17), s menšími modifikacemi. Krátce, pro first-strand cDNA syntézu, 4 µL 5× first-strand pufru (Invitrogen), 1 µL 0,1 M dithiothreitolu (DTT), 1 µL 10 mM dntp, 1 µL (40 U) RNaseOUT™ (Invitrogen) a 1 µL vody byly přidány do denaturované RNA/T7-Ne-(dT)18 primer mixure a inkubovány při 37°C po dobu 3 min do nasedají primery. Pak, 2 µL (400 IU) SUPERSCRIPT III RNase H-reverzní transkriptázy (Invitrogen) byly přidány do reakční směsi a teplota byla nastavena na 50°C-start prvních-strand cDNA syntézu. Po 1 h, druhá-strand cDNA syntéza byla provedena, jak bylo dříve popsáno Gubler a Hoffman (18); first-strand cDNA směsi, 91 µL vody, 30 µL 5× za druhé-strand pufru (Invitrogen), 3 µL 10 mM dntp, 1 µL (10 U) Escherichia coli ligázy DNA, 4 µL (40 U) E. coli DNA polymerázy a 1 µL (2 U) E. coli RNase H byly přidány, a směs byla inkubována při teplotě 16°C po dobu 2 h. cDNA termini pak byly end-leštěné s 10 U T4 DNA polymerase (Invitrogen) při 16°C po dobu 5 min, a reakce byla zastavena přidáním 10 µL 0,5 M ethylendiamin ethylendiamintetraoctové kyseliny (EDTA). Výsledný cDNAs byli čistí extrakcí fenol/chloroform/isoamyl alcohol (25:24:1), následuje ethanol srážek jak již bylo dříve popsáno (17), s výjimkou pro použití 1 µg kvasinkovou tRNA místo polyadenylic kyseliny jako nosič. Vysrážený cDNA byl rozpuštěn v 50 µL TE (10 mM Tris-HCl, pH 8.0, a 1 mM EDTA), ve směsi s 30 µL 20% (w/v) polyethylenglykol (PEG) 6000/2.5 M NaCl roztoku, a pak inkubovány na ledu po dobu 1 h (17). Po inkubaci byla směs cDNA centrifugována při 18 000× g při 4°C po dobu 15 minut. Výsledná peleta cDNA byla dvakrát propláchnuta 70% ethanolem, vysušena a poté resuspendována ve 30 µL vody. Purifikované cDNA byly ligovány 500 pmol SP6 adaptorem (Tabulka 1) za použití 5 U T4 DNA ligázy (Invitrogen)v 50 µL reakčním objemu při 16°C přes noc. Adapter-ligovaná směs byla zředěna dvakrát vodou a potom byly cDNA čištěny pomocí kolony DNAclear™ (Ambion, Austin, TX, USA). Eluát (v 16 µL vody) byl použit jako šablona pro syntézu cRNA. Pomocí MEGAscript® T7 kit (Ambion) byly Crna syntetizovány při 37°C přes noc v reakčním objemu 40 µL. Poté, co byly templátové cDNA degradovány 4 U Dnázou I, byly syntetizované Crna čištěny pomocí Mini Kit RNeasy® (QIAGEN, Valencia, CA, USA) a eluovány 100 µL vody. Koncentrace cRNA byla stanovena ultrafialovou (UV) absorpcí. Pro druhé kolo cRNA zesílení, 2 µg syntetizován cRNA a 100 pmol SP6 UP primer (rovná horní-pramenné oligonukleotid z SP6 adaptér je uvedeno v Tabulce 1) byly použity jako šablony a základní nátěr, respektive. Syntéza cDNA ve druhém kole byla provedena jako v původní reverzní transkripci ze zdrojové RNA, kromě toho, že žíhání primeru SP6 UP bylo provedeno při 50°C po dobu 3 minut. Po purifikaci získané dvouvláknové cDNA Dnakleární kolonou bylo jako šablona pro další amplifikaci cRNA s podporou SP6 RNA použito 0,5 µg cDNA. Pomocí MEGAscript SP6 kit (Ambion), cRNA syntéza byla provedena při teplotě 37°C po dobu 6 h. Po templátové cDNA byl degradován pomocí Dnázy I, integrovaný cRNAs byly čištěny, jak je popsáno výše.
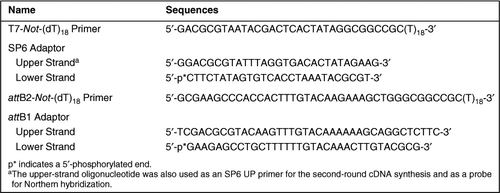
cDNA Knihovna Výstavby
Dvě mikrogramů výsledné cRNAs byly podrobeny double-stranded cDNA syntéza pomocí 100 pmol attB2-Ne-(dT)18 primeru (Tabulka 1). Kroky syntézy cDNA, otupení, čištění a ligace adaptérů byly provedeny stejně jako v předchozích syntézách cDNA, kromě toho, že adaptér attB1 (Tabulka 1 500 pmol) byl ligován na dvouvláknové cDNA. Po attB1 adaptér-ligované cDNAs byl čištěn po sobě jdoucích ošetření fenol/chloroform/isoamyl alkohol extrakce, ethanol srážek, a PEG/NaCl srážek v tomto pořadí, byly rozpuštěny v 50 µL TE a zacházet s RNase A (na konečnou koncentraci 10 µg/mL, Invitrogen) k degradaci kontaminovaných RNA při 37°C po dobu 30 min. Reakční směs byla opět purifikována extrakcí fenol / chloroform/isoamylalkoholem, následovanou srážením ethanolu a srážením PEG/NaCl. Výsledné cDNA byly rozpuštěny v 15 µL TE a jejich množství bylo odhadnuto z intenzity fluorescenčního barvení po elektroforéze na agarózovém gelu (4). Na attB1-ligován cDNAs (36 ng) byla podrobena in vitro rekombinace reakce (Gateway® Systém; Invitrogen) s 250 ng attP-pSP73 dárce vektoru, jak bylo popsáno dříve (4,16,17). Stručně řečeno, attB1-ligován cDNA a attP-pSP73 dárce vektorové byly promíchány a inkubovány v 10 µL reakční objem obsahující 2 µL 5× BP Clonase™ reaction pufru a 2 µL BP Clonase (oba z Invitrogen) při 25°C přes noc. Na cDNA směs se smísí s 2 µg proteinázy K (Invitrogen) při 37°C po dobu 10 min, aby se reakce zastavila a extrahována fenol/chloroform/isoamyl alkohol, následuje ethanol srážek s 2 µg kvasinkovou tRNA jako dopravce. Vysrážená cDNA byla rozpuštěna v 10 µL vody a 1 µL směsi bylo použito pro transformaci buněk E. coli ElectroMAX™ DH10B™ (Invitrogen). Po titraci výsledné knihovny cDNA byly plazmidy cDNA získány z přibližně 1 000 000 kolonií metodou alkalického dodecylsulfátu sodného (SDS), jak bylo dříve popsáno (4,19). Výsledné klony cDNA byly ve formě plazmidu nesoucího attL místa. Protože plazmidy nesoucí attB místa jsou v některých aplikacích vyžadovány, byly tyto plazmidy převedeny na plazmidy nesoucí attB místa, jak bylo dříve popsáno (16). Tato cDNA knihovna byla následně označena jako MB-AL (knihovna odvozená od Crna s amplifikovaným mozkem myši).
stejným způsobem, jak je popsáno pro MB-AL, jsme zkonstruovali knihovnu cDNA z neamplifikované RNA (100 µg celkové RNA myšího mozku) a označili ji jako MB-CL(konvenčně konstruovaná knihovna myšího mozku).
Zkoumání Velikost-Bias Vliv na Zesílen cRNAs
zkoumat velikost-bias vliv na cRNAs, v prvním kole cRNAs přepisována pomocí T7 RNA polymerázy a druhý-kolo cRNAs přepsal SP6 RNA polymerázy byly prováděny na 1.0% agarózovém gelu (1 µg) a pak přeneseny na nylonovou membránu (Biodyne® B Nylonové Membrány; PALL, East Hills, NY, USA). Byly hybridizovány s 32P-značené SP6 UP primer (Tabulka 1) a Ne-(dT)18 (5′-GCGGCCGCTTTTTTTTTTTTTTTTTT-3′) oligonukleotid, resp. Deset pikomolů každé sondy bylo označeno ATP (přibližně 3000 Ci / mmol; Amersham Biosciences, Piscataway, NJ, USA) pomocí polynukleotid kinázy T4 (Takara, Kjóto Japonsko). Po noční hybridizaci v GMC pufru (20) při 45°C byly tyto membrány extenzivně promyty v 1× standardním fyziologickém citrátu (SSC) / 1% roztoku SDS třikrát při pokojové teplotě. Analýza hybridizovaných signálů byla provedena na analyzátoru obrazu BAS 2000 (Fuji Photo Film, Tokio, Japonsko)
vyhodnocení knihoven cDNA
gelová elektroforéza. Distribuce velikostí vložek cDNA byla zkoumána elektroforézou Crna syntetizovaných T7 RNA polymerázou za použití NotI-štěpených MB-AL A MB-CL plazmidových cDNA jako šablon. Po čištění pomocí Mini soupravy RNeasy byly Crna elektroforovány na agarózovém gelu obsahujícím formaldehyd s dokonalými RNA markery (EMD Biosciences, Madison, WI, USA). Po obarvení gelu SYBR® Green II (Invitrogen) byla intenzita fluorescenčního barvení analyzována pomocí softwaru ImageQuant® (verze 5.0) na FluorImager 595 (Amersham Biosciences).
náhodná jednoprůchodová sekvenční analýza. 3 ‚ – koncové sekvence cDNA klonů byly zkoumány sekvenováním DNA. Plasmidová DNA z 768 náhodně vybral klony byly čištěny pomocí MFX-9600 Magnia® (Toyobo, Osaka, Japonsko) a analyzovány pomocí RISO 384-Kapilární Sekvencer (Shimadzu, Kyoto, Japonsko) (16). Po oříznutí vektorové sekvence pomocí Sequencher™ Software verze 4.1 (Hitachi Software, Tokio, Japonsko), získané cDNA sekvence, které byly delší než 200 nukleotidů zbytky byly clustery s cDNA záznamů v GenBank® databáze a naše in-house mouse databáze. Vyšetření integrity 3′ koncích cDNA klonů byla provedena analýza toho, zda je nebo není kanonický polyadenylation signál hexamer (5′-AATAAA-3‘) nebo jeho single-základní varianty (11 signál hexamer) byly nalezeny v 50 nukleotidových zbytků proti proudu od poly(A) tail z cDNAs (21).
cDNA microarray. Byly provedeny analýzy Microarray k porovnání populací cDNA v knihovnách MB-AL A MB-CL. Byly připraveny domácí cDNA nylonové microarrays s 3534 sondami a byla provedena radioizotopická detekce (22). Krátce, cDNA plazmidů, které byly izolovány v našem ústavu a pro které je plné sekvencí nebo na konci sekvence již byly známy, byly spatřeny pomocí GeneTAC™ RA1 (Genomic Solutions, Ann Arbor, MI, USA) na Biodyne B nylonové membrány, a pak stanovena následující pokyny výrobce. Seznam cDNA imobilizovaných na tomto microarray je k dispozici na vyžádání od autorů. MB-AL A MB-CL cílové cDNA byly připraveny pomocí reverzní transkripce z každého z 1 µg vzorků cRNA použitých pro analýzu velikosti vložky, jak je popsáno výše. Tyto cíle byly syntetizovány a označeny v reakční směsi obsahující 1 µg každého cRNA, 100 ng random hexamer, 1× first-strand pufru, 10 mM DTT, každý 800 µM dATP, dTTP a dGTP, 800 nM, dCTP, 5 µL dCTP (>2500 Ci/mmol; Amersham Biosciences) a 100 U horní Index II RNase H – reverzní transkriptázy při pokojové teplotě po dobu 10 min a pak na 42°C po dobu 1 h. Po alkalické lýze a neutralizace, označené cDNAs byly čištěny pomocí QIAquick® PCR purification kit (QIAGEN) a počítal. Cílové cDNA byla podrobena hybridizace s cDNA nylon microarray v přítomnosti 1 µg polyadenylic kyseliny a 2,5 µg Myši Cot-1 DNA® (Invitrogen) v 250 µL PerfectHyb™ vyrovnávací paměť (Toyobo) při 68°C přes noc. Po přísné mytí při 68°C s 2× SSC/1% SDS (dva myje 15 min každý) a pak s 0,1 x SSC/1% SDS (dva myje 30 min každý), hybridizační signály byly zjištěny a analyzovány na FLA-8000 (Fuji Photo Film). Byly vybrány skvrny cDNA vykazující silnější signály než signály kontroly pozadí a podrobeny další analýze. Po provedení globální normalizace byly získány rozptylové grafy intenzity signálu cDNA skvrn pocházejících z cílů MB-AL A MB-CL.
RNA blot hybridizace. Hybridizace RNA blot byla provedena za účelem zkoumání účinků zkreslení velikosti. MB-AL a MB-CL Crna (každá 1,5 µg, syntetizovaná, jak je popsáno výše) byly elektroforovány na agarózovém gelu obsahujícím formaldehyd a přeneseny na nylonovou membránu Biodyne B. CDNA sondy byly připraveny pomocí PCR amplifikace. Primery byly navrženy na základě sekvencí registrovaných v databázi GenBank nebo v naší interní databázi; šablony byly plazmidy cDNA, které jsme izolovali. Sonda ribozomální S6 protein byl 724 bp v délce zesílen s 5′-CGCTCGGCTGTGTCAAGATG-3′ a 5′-GAGGACAGCCTACGTCTCTTGG-3′ primery a sondy tepelného šoku protein (hsp) 70, 940 bp na délku, byl zesílen s 5′-GATGGACAAGGCGCAGATCC-3′ a 5′-CTCGATGGTGGGTCCTGAGC-3′ primery. Tyto sondy byly označeny dCTP (přibližně 3000 Ci / mmol; Amersham Biosciences) pomocí systému značení dna RadPrime (Invitrogen). Po noční hybridizaci v PerfectHyb pufru při 65°C byly membrány promyty postupně 0,1× SSC/1% SDS při pokojové teplotě po dobu 5 minut a po dobu 15 minut a poté při 65°C po dobu 30 minut. Hybridizační signály byly detekovány na analyzátoru obrazu BAS 2000.
Výsledky a diskuse
cDNA knihovna stavební metody, které jsme vyvinuli v této studii je ukázáno na Obrázku 1 (nově zavedené kroky jsou označeny hvězdičkou). Tato metoda se skládá ze dvou kol kroku amplifikace cRNA, přeměny Crna na cDNA a rekombinačního klonování na plazmid. Klíčovým krokem v této metodě je ligace adaptéru k označení cDNA končí sekvencí promotoru SP6. Jakmile můžeme úspěšně syntetizovat cDNA v tomto formátu, je možné specificky reverzní transkribovat Crna prvního kola, které obsahují sekvenci promotoru SP6 na jejich 3 ‚ konci. Crna druhého kola jsou syntetizovány SP6 RNA polymerázou za použití výsledných dvouvláknových cDNA jako šablon. Po dvou kolech RNA polymeráza-asistované amplifikaci, výsledný cRNAs jsou pak převedeny na double-stranded cDNAs reverzní transkripce pomocí attB2-Ne-(dT)18 primer. Použití attB2-Ne-(dT)18 primer v reverzní transkripci nám umožňuje převést pouze cRNAs nesoucí sekvenci 5′-(A)18GCGGCCGC-3 na svých 3‘ koncích. Protože tyto modifikace by měly umožňovat pouze amplifikaci a klonování cDNA, které mají správně označené konce, očekáváme, že tato metoda minimalizuje zkreslení velikosti během amplifikace podporované RNA polymerázou.

jsou znázorněny kroky v amplifikaci Crna s pomocí Adapter-ligation a následující konstrukci knihovny cDNA. Abychom udrželi amplifikované velikosti cDNA s velikostmi cDNA prvního kola, zavedli jsme některé modifikace v konvenčních metodách podporovaných RNA polymerázou (zvýrazněné hvězdami). Modré a červené čáry označují cDNA a RNA. E. coli, Escherichia coli.
Chcete-li vyzkoušet účinnost metody, postavili jsme cDNA knihovny konvenční metodou (16) pomocí nonamplified celkové RNA z myší mozek (100 µg) a výše popsanou metodou pomocí 1 µg celkové RNA z myší mozek. Tabulka 2 shrnuje množství cDNA a cRNA získané v každém kroku, vypočteno jako průměr tří nezávislých experimentálních běhů konstrukce MB-AL. Jako konečný výstup jsme získali průměrně 6,4×107 nezávislých klonů cDNA. Protože tento počet cDNA klonů pochází z celkem 36 ng cDNA vytvořené pomocí amplifikační protokol, můžeme vypočítat, že tato metoda dává přibližně 1.2×1011 cDNA klonů z 1 µg celkové RNA.

zkoumat velikost-zkreslení efekt během amplifikační kroky experimentálně, dále jsme porovnávali velikost distribuce v prvním kole a druhé kolo cRNA RNA blot hybridizace analýzy. V těchto experimentech, v prvním kole a druhé kolo cRNAs byly hybridizovány s SP6 UP primer a s Ne-(dT)18 oligonukleotid, v tomto pořadí, protože tyto oligonukleotidových sond detekovat pouze cRNAs správně přepsány do konce. Jak je znázorněno na obrázku 2, bylo zjištěno, že velikost RNA na vrcholu hybridizačního signálu v cRNA druhého kola je téměř stejná jako v cRNA prvního kola, ale s mírným posunem na menší velikost. Máme podezření, že ke zkrácení Crna pravděpodobně došlo během syntézy cDNA ve druhém kole pomocí primeru SP6 UP.

byla provedena hybridizace RNA blot za účelem zkoumání vlivu zkreslení velikosti na Crna. Byly vyneseny hybridizační signály ve směru elektroforézy cRNA. Velikost RNA byla stanovena na základě mobility markeru žebříku RNA. Modrá čára, první kolo Crna hybridizované s SP6 UP sondou; červená čára, druhé kolo cRNA hybridizované s ne – (dT)18 sondou; hodnota PSL, intenzita signálu indikovaná libovolně softwarem Image Gauge (Fuji Photo Film).
dále posoudit kvalitu výsledné amplifikované cDNA knihovny, také jsme porovnávali různé funkce cDNA knihovny vytvořené s a bez zesílení (MB-AL a MB-CL, respektive). Nejprve jsme zkoumali distribuci velikosti cDNA vložky, jak je znázorněno na Obrázku 3A. Výsledky fluorescenční intenzity signálů získaných z gelu-barvení obraz je uvedeno, že cDNA-li vložit velikostí MB-AL a MB-CL byly podobné, ale nejvyšší bod byla o něco menší v MB-AL (0.65 kb) ve srovnání s, že v MB-CL (0.8 kb). Dále jsme analyzovali 3 ‚ – koncové sekvence náhodně vzorkovaných klonů cDNA z každé knihovny. Shlukování výsledků pro tyto vyjádřené pořadí značek (Est) jsou uvedeny na Obrázku 3B a naznačují, že složitost MB-AL byla stejně vysoká jako MB-CL. Je však třeba poznamenat, že v MB-AL zmizely vysoce redundantní klony cDNA. Pomocí statistické analýzy (Chí test) bylo prokázáno, že rozdíl mezi propouštěním MB-AL a MB-CL byl významný (pravděpodobnost, že propouštění v MB-AL a MB-CL jsou podobné, je <0.001). Kromě toho jsme zkoumali integritu 3‘ konců cDNA analýzou, zda každý est obsahoval hexamer polyadenylačního signálu nebo ne. Výsledky ukázaly, že 80,1% a 87,3% cDNA klonů v MB-AL A MB-CL obsahovaly věrohodné polyadenylační signální sekvence (21). Snížení frekvence výskytu polyadenylation signál hexamer v MB-AL pravděpodobně indikován nárůst počtu cDNA klonů, kde cDNA syntéza byla vnitřně naplněn v důsledku dvou kolech dT připravovat.

MB-AL (myš brain amplified cRNA-derived library) byla hodnocena ve srovnání s konvenčně konstruovanou knihovnou, MB-CL. A) porovnání délek vloženého cDNA. In vitro transkribované Crna byly velikostně frakcionovány na agarózovém gelu a obarveny. Jejich fluorescenční intenzity ve směru elektroforézy (označené jako velikost RNA) jsou uvedeny v relativních fluorescenčních jednotkách (RFU). (B) shlukování výsledků MB-AL A MB-CL 3 ‚ – koncové sekvence(ESTs). C) srovnání populace cDNA v MB-AL A MB-CL. Signály získané hybridizací microarray byly vyneseny. (D) výsledky hybridizace RNA blot o ribozomálním proteinu S6 a HSP 70. MB-AL cRNAs byly v levém pruhu a MB-CL byly v pravém pruhu (označené AL a CL). Šipky označují pozice předpokládané velikosti RNA každého genu. hsp, protein tepelného šoku.
microarray hybridizací jsme zkoumali populaci cDNA MB-AL ve srovnání s populací MB-CL. Obrázek 3C ukazuje rozptylový graf hybridizačních signálů cílů MB-AL A MB-CL. Výsledky ukázaly značně vysoká korelace hybridizačních signálů mezi MB-AL a MB-CL cíle, což naznačuje, že tam nebyl nepříznivý účinek zesílení v cDNA populace.
nakonec jsme provedli analýzu RNA blot hybridizace, abychom zkoumali účinky zkreslení velikosti s důrazem na geny domácnosti (obrázek 3D). U ribozomálního proteinu S6 a hsp 70 byly velikosti cRNA v MB-AL i MB-CL podobné a srovnatelné s těmi, které se očekávají z jejich hlášených nukleotidových sekvencí. Nicméně, hybridizační vzor hsp 70 Crna v MB-AL byl mírně odlišný od vzoru v MB-CL, což naznačuje, že efekt zkreslení velikosti nebyl zcela odstraněn.
metoda konstrukce knihovny cDNA popsaná v této zprávě byla navržena tak, aby minimalizovala zkreslení velikosti během amplifikačních kroků podporovaných RNA polymerázou. Taková RNA polymeráza-asistované knihovna stavební metody z malé RNA bazén pro komplexní sekvenování analýza nebyla dobře studoval, zatímco cDNA/cRNA knihovna stavební metody microarray hybridization byly aktivně prováděny. Ačkoli Lukyanov et al. (23) a Piao et al. (24) uvádí metody pomocí PCR amplifikace pro konstrukci cDNA knihovna z submicrogram množství celkové RNA, PCR amplifikace vnitřně má své nevýhody, jako jsou závažné velikosti a počtu obyvatel zkreslení a nízká věrnost cDNA amplifikace. Proto jsme se v této studii pokusili vyvinout metodu konstrukce knihovny cDNA založenou na amplifikaci podporované RNA polymerázou.
pro tento účel jsme navrhli novou strategii amplifikace pomocí RNA polymerázy, jak je znázorněno na obrázku 1, protože metody Eberwine et al. (5), Huang et al. (10)A Lin et al. (11) stále mají nevýhody pro přípravu cDNA obsahujícího plazmid vhodné pro komplexní analýzu genové struktury. V prvním případě se syntéza cDNA po amplifikaci cRNA provádí náhodnými hexamery,což činí amplifikované cDNA nevyhnutelně kratší než původní (5,7). V druhé, zavedení homopolymeric ocas na first-strand cDNA konci terminal deoxynucleotidyl transferase (TdT) nebo Moloney murine leukemia virus (MMLV) reverzní transkriptázy činnosti (10,11) dává smysl, aby se minimalizovalo velikosti-zkreslení efekt během RNA polymeráza-asistované zesílení jako v naší metodou. Je však známo, že homopolymerní způsob ocasu způsobuje zkrácení cDNA v důsledku neočekávaného primingu syntézy cDNA z homopolymerního úseku vnitřních RNA sekvencí. Protože metoda Adapter-ligation popsaná v této studii by mohla poskytnout specifickou sekvenci pro priming cDNA, mohli bychom očekávat snížení rozsahu zkrácení cDNA pomocí naší metody.
v zásadě by naše metoda měla amplifikovat pouze cDNA lemované vnitřním poly(a) ocasem a sekvencí adaptérů SP6, které byly generovány při syntéze cDNA v prvním kole. Výsledky znázorněné na obrázku 2 a obrázek 3A byly v podstatě v souladu s tím, i když populace cDNA v menším rozsahu byla mírně zvýšena. Výsledky analýzy RNA blot (obrázek 3D) také ukázaly, že efekt zkreslení velikosti by mohl být snížen, ale ne zcela odstraněn ani v naší metodě. To bylo pravděpodobně proto, že zkrácené cDNA připojené ocasem poly(A) a sekvencí promotoru SP6 byly generovány během konverze cRNA na cDNA. Tento názor byl podpořen skutečností, že výskyt sekvencí polyadenylačního signálu v MB-AL byl významně nižší než výskyt pozorovaný v konvenční knihovně cDNA (21). Zkrácení cDNA by mohlo nastat během syntézy cDNA v důsledku vnitřního primingu RNA primerem dT-tailed a primerem SP6. Protože homopolymeric táhne se často vyskytují v eukaryotických mRNAs, homopolymeric odkalovacích metoda může ukládat přísnější velikost zkreslení na více-kolo cRNA zesílení než náš adaptér-ligační metoda. Ačkoli zvýšení reakční teploty během reverzní transkripce a snížení koncentrace primeru by mohlo do jisté míry potlačit aberantní vnitřní priming v syntéze cDNA, je známo, že je obtížné to v současné době zcela eliminovat. Ačkoli jsme tedy provedli dvoukolovou amplifikaci cRNA pro demonstraci celkového protokolu, v praxi doporučujeme přeskočit amplifikaci Crna druhého kola, pokud lze získat dostatek cRNA v reakci prvního kola.
vzhledem k těmto výsledkům jsme dospěli k závěru, že naše nová metoda je užitečná pro konstrukci knihovny cDNA z malého množství počáteční RNA. Ve skutečnosti jsme úspěšně a rutinně použili tuto metodu pro konstrukci knihoven cDNA, když je množství celkové RNA menší než 1 µg(data nejsou zobrazena). Protože 1 µg celkové RNA by mohly být získány z 105-106 buněk savců nebo 1 mg savčích tkání, cDNA knihovny mohou být snadno vyrobeny z buněk, frakcionaci mobilní třídiče nebo microdissection pomocí naší metody. Kromě toho, protože počítáme, že tato metoda nám umožňuje získat více než 105 cDNA klonů z 1 pg celkové RNA, která je v blízkosti, aby celkové množství RNA v jedné buňce, jedné buňce-odvozena cDNA knihovna mohla být postavena na základě této adaptér-podvázání-asistované cRNA zesílení.
Děkujeme
autoři jsou vděční za poučení statistické analýzy od Dr. Kazuharu Misawa; rozdělení plasmidových Dna od Dr. Hisashi Koga; a technickou pomoc od Paní Akiko Ukigai-Ando a Pan Takashi Watanabe. Studie byla podporována grantem z Kazusa DNA Research Institute R. O., R. F. K. a O. O.
Konkurenční Zájmy Prohlášení
autoři prohlašují, žádné konkurenční zájmy.
- 1. Carninci, P., C. Kvam, a. Kitamura, T. Ohsumi, y. Okazaki, m. Itoh, m. Kamiya, k. Shibata, et al.. 1996. Vysoce účinné klonování cDNA v plné délce biotinylovaným Cap trapperem. Genomika 37: 327-336.Crossref, Medline, CAS, Google Scholar
- 2. Ohara, o., T. Nagase, k. Išikawa, D. Nakajima, m. Ohira, N. Seki a N. Nomura. 1997. Konstrukce a charakterizace cDNA knihoven lidského mozku vhodných pro analýzu cDNA klonů kódujících relativně velké proteiny. DNA Res.4:53-59.Crossref, Medline, CAS, Google Scholar
- 3. Suzuki, Y., K. Jošitomo-Nakagawa, k. Maruyama, a. Suyama a s. Sugano. 1997. Konstrukce a charakterizace knihovny cDNA obohacené o celou délku a obohacené o 5′. Gene 200: 1149-1156.Crossref, Google Scholar
- 4. Ohara, O.A G. Temple. 2001. Směrová konstrukce knihovny cDNA podporovaná rekombinační reakcí in vitro. Nukleové kyseliny rez.29:e22.Crossref, Medline, CAS, Google Scholar
- 5. Eberwine, J., H. Yeh, k. Miyashiro, y. Cao, s. Nair, R. Finnel, m. Zettel a P. Coleman. 1992. Analýza genové exprese v jednotlivých živých neuronech. Proc. Natle. Acad. Věda. USA 89: 3010-3014.Crossref, Medline, CAS, Google Scholar
- 6. Wang, E., L. D. Miller, G. a. Ohnmacht, E. T. Liu, a F. M. Marincola. 2000. Vysoce věrná amplifikace mRNA pro profilování genů. Adresa. Biotechnol. 18:457–459.Crossref, Medline, CAS, Google Scholar
- 7. Baugh, L. R., a. a. Hill, E. L. Brown, a. C. P. Hunter. 2001. Kvantitativní analýza amplifikace mRNA in vitro transkripcí. Nukleové kyseliny rez.29:e29.Crossref, Medline, CAS, Google Scholar
- 8. Zhumabayeva, B., L. Diatchenko, a. Chenchik a P. D. Siebert. 2001. Použití cDNA generované SMART™pro studie genové exprese u více lidských nádorů. BioTechniques 30: 158-163.Odkaz, CAS, Google Scholar
- 9. Gomes, L., R. Silva, B. S. Stolf, E. B. Cristo, R. Hirata, Jr., F. a. Soares, L. Reis, E. J. Neves, et al.. 2003. Srovnávací analýza amplifikované a neamplifikované RNA pro hybridizaci v cDNA microarray. Anální. Biochem. 321:244–251.Crossref, Medline, CAS, Google Scholar
- 10. Huang, J., T. Lin, D. Chang, s. Lin a s. Ying. 2003. Zkrácený Bcl-2, potenciální premetastický marker u rakoviny prostaty. Biochem. Biophys. Res.Commun. 306:912–917.Crossref, Medline, CAS, Google Scholar
- 11. Lin, S. L. A S. Y. Ying. 2003. konstrukce knihovny mRNA / cDNA pomocí cyklické reakce RNA-polymerázy. Metody Mol. Biol. 221:129–143.Medline, CAS, Google Scholar
- 12. Matz, M.V. 2003. Amplifikace reprezentativních bazénů cDNA z mikroskopických množství živočišné tkáně. Metody Mol. Biol. 221:103–116.Medline, CAS, Google Scholar
- 13. Petalidis, L., S. Bhattacharyya, G. a. Morris, V. P. Collins, T. C. Freeman, a. P. A. Lyons. 2003. Globální amplifikace mRNA pomocí PCR s přepínáním šablon: linearita a aplikace na microarray analýzu. Nukleové kyseliny rez.22:e142.Crossref, Google Scholar
- 14. Poláček, d. c., A. G. Passerini, C. Shi, N. M. Francesco, E. Manduchi, G. R. Grant, S. Powell, H. Bischof, et al.. 2003. Věrnost a zvýšená citlivost diferenciálních transkripčních profilů po lineární amplifikaci nanogramových množství endotelové mRNA. Fyziol. Genomika 13: 147-156.Crossref, Medline, CAS, Google Scholar
- 15. Wilson, C., S. D. Pepper, y. Hey a C. J. Miller. 2004. Amplifikační protokoly zavádějí systematické, ale reprodukovatelné chyby do studií genové exprese. BioTechniques 36: 498-506.Odkaz, CAS, Google Scholar
- 16. Ohara, O., T. Nagase, G. Mitsui, h. Kohga, R. Kikuno, s. Hiraoka, y. Takahashi, s. Kitajima, et al.. 2002. Charakterizace velikostně frakcionované cDNA knihovny generované in vitro rekombinační metodou. DNA Res.9:47-57.Crossref, Medline, CAS, Google Scholar
- 17. Ohara, O.2003. Konstrukce velikostně frakcionované cDNA knihovny za pomoci rekombinační reakce in vitro. Metody Mol. Biol. 221:59–71.Medline, CAS, Google Scholar
- 18. Gubler, U. a B. J. Hoffman. 1983. Jednoduchá a velmi efektivní metoda pro generování cDNA knihoven. Gene 25: 263-269.Crossref, Medline, CAS, Google Scholar
- 19. Sambrook, J. A D.W. Russell. 2001. Příprava plazmidové DNA alkalickou lýzou s SDS, s. 1.31-1.42. Molekulární klonování, (3. vydání). Csh Laboratory Press, Cold Spring Harbor, NY.Google Scholar
- 20. Church, G. M.A W. Gilbert. 1984. Genomické sekvenování. Proc. Natle. Acad. Věda. USA 81: 1991-1995.Crossref, Medline, CAS, Google Scholar
- 21. Beaudoing, E., S. Freier, J. R. Wyatt, J. M. Claverie, a. D. Gautheret. 2000. Vzory využití variantního polyadenylačního signálu v lidských genech. Genome Res.10:1001-1010.Crossref, Medline, CAS, Google Scholar
- 22. Tanaka, T. S., S. a. Džarádáta, M. K. Lim, G. J. Kargul, X. Wang, M. J. Grahovac, S. Pantano, Y Sano, et al.. 2000. Profilování exprese v celém genomu placenty a embrya v polovině těhotenství pomocí 15 000 myších vývojových cDNA microarray. Proc. Natle. Acad. Věda. USA 97: 9127-9132.Crossref, Medline, Google Scholar
- 23. Lukyanov, K., L. Diatchenko, a. Chenchik, a. Nanisetti, P. Siebert, N. Usman, m. Matz a s. Lukyanov. 1997. Konstrukce cDNA knihoven z malého množství celkové RNA pomocí supresního PCR efektu. Biochem. Biophys. Res.Commun. 230:285–288.Crossref, Medline, CAS, Google Scholar
- 24. Piao, Y., N. T. Ko, m. k. Lim a M. Ko. 2001. Konstrukce long-transkriptů obohacených cDNA knihoven z submikrogramových množství celkových RNA univerzální metodou PCR amplifikace. Genome Res.11:1553-1558.Crossref, Medline, CAS, Google Scholar
